[ad_1]
Zhang, Y. & Harrison, T. Gigantopithecus blacki: un singe géant du Pléistocène d’Asie revisité. Un m. J. Phys. Anthropol. 162 (Suppl. 63), 153–177 (2017).
Harrison, T. Apes parmi les branches enchevêtrées des origines humaines. Science 327532-534 (2010).
Begun, D. R. Comment identifier (par opposition à définir) une homoplasie: exemples de grands singes fossiles et vivants. J. Hum. Evol. 52559-572 (2007).
Kelley, J. dans Le record fossile de fossiles (ed. Hartwig, W. C.) 369–384 (Cambridge Univ. Press, 2002).
Miller, S. F., White, J. L. et Ciochon, R. L. Évaluation de la variation de forme mandibulaire au sein Gigantopithecus en utilisant une approche morphométrique géométrique. Un m. J. Phys. Anthropol. 137, 201-212 (2008).
Grehan, J.R. & Schwartz, J.H. Evolution du deuxième orang-outan: phylogénie et biogéographie d'origines d'hominidés. J. Biogeogr. 36, 1823-1844 (2009).
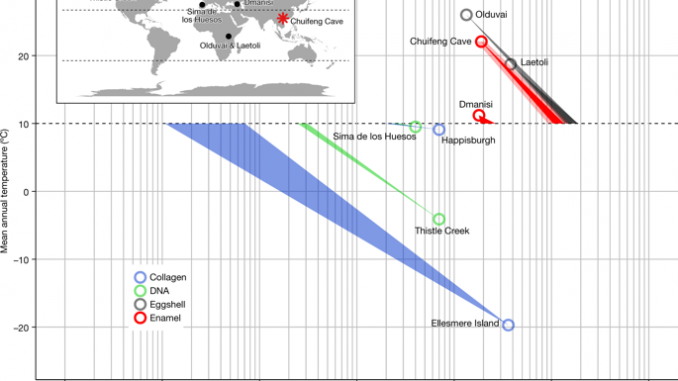
Shao, Q. et al. ESR, série U et datation paléomagnétique de Gigantopithecus faune de la grotte de Chuifeng, Guangxi, sud de la Chine. Quat. Res. 82270-280 (2014).
Wang, W. Nouvelles découvertes de Gigantopithecus blacki dents de la grotte de Chuifeng dans le bassin Bubing, Guangxi, sud de la Chine. J. Hum. Evol. 57, 229-240 (2009).
Bartlett, J. D. et al. Interactions protéine-protéine de la matrice d'émail en développement. Curr. Haut. Dev. Biol. 74, 57-115 (2006).
Dean, M. C. & Schrenk, F. Épaisseur de l'émail et développement dans une troisième molaire permanente de Gigantopithecus blacki. J. Hum. Evol. 45381 à 388 (2003).
Von Koenigswald, G.H.R. Eine fossile Saugetierfauna mit Simia aus Sudchina. Proc. K. Ned. Akad. Humide. 38, 872 à 879 (1935).
Zhang, Y. et al. Nouveau 400–320 ka Gigantopithecus blacki Restes de la grotte Hejiang, ville de Chongzuo, Guangxi, sud de la Chine. Quat. Int. 354, 35–45 (2014).
Zhao, L. X. & Zhang, L. Z. Nouvelles preuves fossiles et analyse du régime alimentaire de Gigantopithecus blacki et sa distribution et son extinction dans le sud de la Chine. Quat. Int. 286, 69–74 (2013).
Pei, W. C. Fouilles de Liucheng Gigantopithecus grotte et exploration d'autres grottes à Kwangsi. Mémoire de l'Institut de Paléontologie et de Paléoanthropologie Vertébrée, Académie Sinica sept1-54 (1965).
Bocherens, H. et al. Flexibilité du régime alimentaire et de l'habitat chez les mammifères sud-asiatiques du Pléistocène: implications pour le destin du singe fossile géant Gigantopithecus. Quat. Int. 434148-155 (2017).
Ciochon, R. et al. Co-occurrence datée de l'homo erectus et Gigantopithecus de la grotte Tham Khuyen, Vietnam. Proc. Natl Acad. Sci. Etats-Unis 933016-3020 (1996).
Cappellini, E. et al. Le protéome de l'émail du Pléistocène précoce de Dmanisi se résout Stephanorhinus phylogénie. La nature 574103-107 (2019).
Meyer, M. et al. Séquences d'ADN nucléaire provenant des hominines de la Sima de los Huesos du Pléistocène moyen. La nature 531504–507 (2016).
Wadsworth, C. & Buckley, M. Dégradation du protéome dans les fossiles: étude de la longévité de la survie des protéines dans les os anciens. Commun rapide. Spectrométrie de masse. 28, 605–615 (2014).
Stewart, N. A. et al. L’identification des peptides par nanoLC-MS / MS à partir d’émail dentaire humain à la suite d’une simple extraction par attaque acide. Avances RSC 6, 61673 à 61679 (2016).
Castiblanco, G.A. et al. Identification des protéines de l'émail humain permanent en éruption. EUR. J. Oral Sci. 123, 390–395 (2015).
Cristobal, A. et al. Vers un flux de travail optimisé pour la protéomique intermédiaire. Anal. Chem. 89, 3318 à 3325 (2017).
Cox, J. et Mann, M. MaxQuant permettent des taux d’identification de peptides élevés, des exactitudes de masse individualisées dans la plage p.p.b. et une quantification des protéines à l’échelle du protéome. Nat. Biotechnol. 261367-1372 (2008).
Tagliabracci, V. S. et al. La kinase sécrétée phosphoryle les protéines extracellulaires qui régulent la biominéralisation. Science 3361150-1153 (2012).
Prado-Martinez, J. et al. Diversité des grands singes et histoire de la population. La nature 499471–475 (2013).
Nater, A. et al. Preuves morphométriques, comportementales et génomiques d'une nouvelle espèce d'orang-outan. Curr. Biol. 27, 3487 à 3498 (2017).
Tang, N. & Skibsted, L. H. Liaison de calcium à des acides aminés et à de petits peptides de glycine en solution aqueuse: vers la conception d'un peptide pour une meilleure biodisponibilité du calcium. J. Agric. Food Chem. 644376-4389 (2016).
Heiss, A. et al. Base structurelle de l'inhibition de la calcification par la glycoprotéine alpha 2-HS / la fétuine-A. Formation de particules de calciprotéine colloïdale. J. Biol. Chem. 27813333-13341 (2003).
Demarchi, B. et al. Les séquences protéiques liées aux surfaces minérales persistent dans le temps profond. eLife 5, e17092 (2016).
Price, P. A., D. Toroian et J. Lim. J. E. Minéralisation par exclusion d'inhibiteur: la calcification du collagène avec la fétuine. J. Biol. Chem. 28417092-17101 (2009).
Kono, R. T., Zhang, Y., Jin, C., Takai, M. et Suwa, G. Une évaluation tridimensionnelle de l'épaisseur de l'émail molaire et du schéma de distribution Gigantopithecus blacki. Quat. Int. 354, 46–51 (2014).
Mackie, M. et al. Profil paléoprotéomique des couches de conservation sur une peinture murale italienne du XIVe siècle. Angew. Chem. Int. Ed. 577369–7374 (2018).
Jin, C. et al. Séquence chronologique du début du pléistocène Gigantopithecus faunes de sites de grottes dans la région de Chongzuo, Zuojiang River, Chine du Sud. Quat. Int. 354, 4-14 (2014).
Huang, W.C. R. et al. De bonne heure Homo et artéfacts associés en provenance d’Asie La nature 378275-278 (1995).
Pei, W. Découverte de Gigantopithecus mandibules et autre matériel dans le district de Liucheng, dans le centre de Kwangsi, dans le sud de la Chine. Vertebrata PalAsiatica 165-71 (1957).
Sun, L. et al. Séquence magnétochronologique du Pléistocène inférieur Gigantopithecus faunes de Chongzuo, Guangxi, sud de la Chine. Quat. Int. 354, 15-23 (2014).
Blain, H.-A., Agustí, J., Lordkipanidze, D., Rook, L. et Delfino, M. Contexte paléoclimatique et paléoenvironnemental des hominines du Pléistocène inférieur de Dmanisi (Géorgie, Petit Caucase) déduites de l'assemblage herpétofaune. Quat. Sci. Tour. 105, 136-150 (2014).
Hendy, J. et al. Un guide pour les études de protéines anciennes. Nat. Ecol. Evol. 2791–799 (2018).
Welker, F. Élucidation des effets protéomiques entre espèces lors de l'identification de protéomes osseux chez l'homme et l'hominine par le biais d'une expérience de bioinformatique. BMC Evol. Biol. 1823 (2018).
Welker, F. Paléoprotéomique pour les études sur l'évolution humaine. Quat. Sci. Tour. 190137-147 (2018).
Hanson-Smith, V. & Johnson, A. PhyloBot: portail Web sur la phylogénétique automatisée, la reconstruction de séquences ancestrales et l'exploration de trajectoires de mutation. PLOS Comput. Biol. 12, e1004976 (2016).
Zhang, J. et al. PEAKS DB: Recherche dans la base de données assistée par séquençage de novo pour l'identification de peptides sensible et précise. Mol. Cellule. Protéomique 11M111.010587 (2012).
Welker, F. et al. Des preuves paléoprotéomiques identifient des hominines archaïques associées au Châtelperronien à la Grotte du Renne. Proc. Natl Acad. Sci. Etats-Unis 11311162-11167 (2016).
de Manuel, M. et al. La diversité génomique des chimpanzés révèle un mélange ancien de bonobos. Science 354477–481 (2016).
Mallick, S. et al. Le projet Simons Genome Diversity: 300 génomes de 142 populations différentes. La nature 538201-206 (2016).
Altschul, S.F., Gish, W., W. Miller, Myers, E. W. & Lipman, D. J.. Outil de recherche d'alignement local de base. J. Mol. Biol. 215403 à 410 (1990).
Katoh, K. & Frith, M. C. Ajout de séquences non alignées dans un alignement existant à l'aide de MAFFT et de LAST. Bioinformatique 283144-3146 (2012).
Guindon, S. et al. Nouveaux algorithmes et méthodes d'estimation de la phylogénie du maximum de vraisemblance: évaluation des performances de PhyML 3.0. Syst. Biol. 59307–321 (2010).
Ronquist, F. et al. MrBayes 3.2: inférence phylogénétique bayésienne efficace et choix de modèle dans un grand espace modèle. Syst. Biol. 61539-542 (2012).
Miller, M.A., Pfeiffer, W. & Schwartz, T. in Atelier d'informatique de passerelle (GCE) 1-8 (Nouvelle-Orléans, 2010).
Schliep, K. P. phangorn: analyse phylogénétique chez R. Bioinformatique 27592-593 (2011).
Le, S. Q. & Gascuel, O. Une matrice améliorée de remplacement des acides aminés en général. Mol. Biol. Evol. 25, 1307-1320 (2008).
Besenbacher, S., C. Hvilsom, T. aaafassos-Bonet, Mailund, T. & Schierup, M. H. L'estimation directe des mutations chez les grands singes réconcilie la datation phylogénétique. Nat. Ecol. Evol. 3286-229 (2019).
Müller, T. & Vingron, M. Modélisation du remplacement des acides aminés. J. Comput. Biol. sept761 à 776 (2000).
Langergraber, K.E. et al. Les temps de génération chez les chimpanzés et les gorilles sauvages suggèrent des temps de divergence plus précoces chez les grands singes et l'évolution humaine. Proc. Natl Acad. Sci. Etats-Unis 10915716-15721 (2012).
Katoh, S. et al. Nouvelle contrainte d'âge géologique et paléontologique pour la scission de la lignée gorille-humain. La nature 530215-218 (2016).
Senut, B. et al. Premier hominidé du Miocène (Formation de Lukeino, Kenya). Comptes Rendus Acad. Sci. Planète Terre IIA. Sci. 332137-144 (2001).
Brunet, M. et al. Un nouvel hominidé du Miocène supérieur du Tchad, en Afrique centrale. La nature 418145-151 (2002).
Haile-Selassie, Y. Hominidés du Miocène supérieur du Middle Awash, Ethiopie. La nature 412178-181 (2001).
[ad_2]