[ad_1]
Toute personne qui a vécu sans chauffage central ni climatisation a dû apprendre les bonnes combinaisons d'ouverture de fenêtre, d'allumage de radiateurs ou de réglage des stores pour obtenir la température idéale. Les thermostats modernes éliminent tout cela: vous les réglez une fois et les contrôleurs intégrés se chargent du reste, quels que soient les changements de temps ou le type de maison. La température peut encore varier un peu, mais tant que les radiateurs et les refroidisseurs sont conçus correctement, elle devrait varier autour du point de consigne, plutôt que de simplement atténuer les effets du froid ou de la chaleur.
, Aoki et ses collègues rapportent un système analogue pour les réactions chimiques dans les cellules vivantes. Plus précisément, ils conçoivent un module de réaction dans lequel deux composants se séquestrent et montrent que son ajout à presque tous les réseaux peut forcer la sortie du système à maintenir une valeur précise proportionnelle à un signal d'entrée, de manière robuste. perturbations externes et incertitude des paramètres internes – comportement connu sous le nom d’adaptation parfaite robuste.
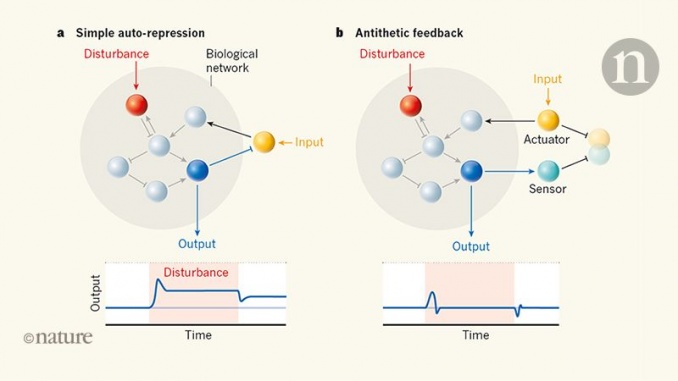
Les résultats sont frappants pour deux raisons. Premièrement, la plupart des circuits biochimiques auto-correctifs atténuent simplement les effets des changements externes, au lieu de les compenser parfaitement. Par exemple, en auto-réprimant leur propre production, les protéines peuvent rendre leurs abondances moins sensibles aux changements de paramètres qu’elles ne le seraient autrement, mais néanmoins dans une certaine mesure (Fig. 1a). Ces systèmes sont donc connus sous le nom de régulateurs homéostatiques car ils maintiennent des niveaux de protéines similaires (homéo) plutôt que identiques (homo). Deuxièmement, l'impact de l'ajout de réactions supplémentaires à un réseau biomoléculaire dépend généralement du contexte. Par exemple, l'ajout d'une étape de répression peut créer une boucle de rétroaction positive ou négative, selon le reste du réseau. La plupart des systèmes ont donc été modélisés et conçus au cas par cas, et il était difficile d’imaginer qu’on puisse trouver un contrôle synthétique universel.
L'approche adoptée par Aoki et ses collègues est aussi frappante que leurs résultats. Quiconque travaille avec des biologistes synthétiques finira par les entendre citer les derniers mots que le physicien Richard Feynman a écrits sur son tableau: «Ce que je ne peux pas créer, je ne le comprends pas.» Cependant, Feynman faisait référence à des dérivations mathématiques plutôt qu'à la construction de réels. Les systèmes mondiaux tels que les réseaux biologiques, et le document d’Aoki et de ses collègues est l’un des rares en biologie synthétique à être à la hauteur de la citation. Les auteurs ont commencé par dériver des règles mathématiques exactes qui s’appliquent à de grandes classes de systèmes de réaction chimique, avant de commencer à construire physiquement des systèmes illustrant la règle.
Bien que les résultats des auteurs portent sur l’abondance moyenne des molécules d’une population de cellules à l’autre, ils ont été calculés à l’aide de cadres qui rendent compte du caractère aléatoire inhérent à des événements de réaction individuels dans des cellules individuelles. Cela peut sembler une distinction subtile: comptabiliser mathématiquement des mécanismes probabilistes, mais ne prédire que les moyennes des distributions statistiques résultantes. Mais pour la plupart des réseaux chimiques, dans lesquels les vitesses de réaction dépendent souvent de manière non linéaire des concentrations, il est nécessaire de prendre en compte les réactions probabilistes même pour prévoir les moyennes correctes. Le niveau de rigueur inhabituel d’Aoki et de ses collègues à cet égard renforce donc considérablement leurs résultats.
Plus spécifiquement, les auteurs se sont concentrés sur une architecture système connue sous le nom de contrôle à rétroaction intégrale antithétique., dans lesquels la rétroaction est mise en œuvre par des molécules d’actionneur et de capteur qui se lient de manière irréversible (Fig. 1b). Si chaque molécule de capteur trouve systématiquement une molécule d'actionneur partenaire, le système détecte que la sortie correspond correctement à l'entrée. Si, au lieu de cela, il y a trop ou trop peu de molécules de capteur, les molécules d'actionneur ajustent automatiquement la production de capteurs pour essayer de trouver le bon équilibre, comme un "système de copain" moléculaire. Aoki et al. prouver mathématiquement non seulement que ce circuit est capable de mettre en œuvre une adaptation parfaite robuste dans tout réseau à réaction chimique, mais également que tous les réseaux présentant une adaptation parfaite robuste doivent à un certain niveau intégrer ce type de motif de rétroaction antithétique.
Les auteurs ont ensuite démontré que leur architecture de contrôle théorique pouvait être mise en œuvre dans des cellules vivantes. Ils se sont concentrés sur un système incorporant des protéines appelées facteurs σ, qui régulent l'initiation de l'expression des gènes chez les bactéries. Certains facteurs σ sont séquestrés par des partenaires de liaison (facteurs anti-σ), tels que le facteur σ SigW de la bactérie Bacillus subtilis et son facteur anti-σ RsiW. Les chercheurs ont intégré SigW dans la bactérie modèle Escherichia coli, et l’a utilisée pour réguler l’expression d’une protéine fluorescente verte (GFP) en tant que rapporteur de l’expression des gènes. Ils ont ensuite couplé l'activation des gènes producteurs de GFP à la production de RsiW, qui séquestrait ensuite SigW. Les niveaux de SigW étaient également régulés par une petite molécule induisant l’expression de la sigW gène, et qui agit comme une entrée dans le circuit. Si le circuit fonctionnait comme prévu, la quantité de fluorescence verte produite par le E. coli les cellules doivent être proportionnelles aux niveaux de SigW, et à l'état d'équilibre doivent être indépendantes de tout autre paramètre.
Bien sûr, Aoki et al. ont montré que la variation de la concentration de l'inducteur pouvait être utilisée pour contrôler la production de GFP comme prévu. Pourtant, lorsque le système a été perturbé par l’ajout d’une enzyme protéase dégradant à la fois la GFP et une protéine affectant la production de RsiW, le signal de fluorescence a été modifié de manière transitoire, puis est revenu à un niveau impossible à distinguer de la valeur initiale, démontrant ainsi que le circuit présente adaptation parfaite. En revanche, dans un système analogue dépourvu de contrôle de rétroaction, la même perturbation abaissait systématiquement la concentration de GFP à environ la moitié de sa valeur initiale. Les auteurs ont même remplacé la GFP par une protéine régulant la croissance cellulaire, produisant ainsi une E. coli souche qui a augmenté à un taux constant, malgré les changements de facteurs qui auraient autrement altéré le taux de croissance.
L’une des orientations futures possibles pour ce type de travail consiste à étudier le circuit dans des cellules uniques, plutôt que ses effets moyens dans différentes populations. D'une part, les travaux récents suggère que des circuits de ce type pourraient augmenter les fluctuations spontanées, comme cela a également été rapporté pour les classes de schéma de réaction associées. D'autre part, des travaux théoriques déjà publiés du même groupe de recherche que celui d'Aoki et al. suggère que des architectures de circuits plus complexes pourraient présenter une adaptation parfaite robuste sans amplifier les fluctuations spontanées. Un tel comportement sera nécessaire pour garantir que les circuits puissent exécuter des fonctions quantitatives précises dans une cellule donnée, malgré le bruit et l'incertitude inhérents. De la même manière que la réduction des taux d’erreur dans les circuits numériques était essentielle au développement des ordinateurs modernes, la capacité de concevoir des sous-réseaux de circuits cellulaires fonctionnant avec précision et robustesse sera probablement nécessaire, alors que nous cherchons à assembler des systèmes cellulaires synthétiques complexes. ceux trouvés dans la nature.
Inscrivez-vous pour le quotidien Briefing Nature Courriel
Restez au courant de ce qui compte dans la science et pourquoi, choisi à la main La nature et d'autres publications dans le monde.
S'inscrire
[ad_2]