[ad_1]
Notre matériel génétique est jonché de séquences d’ADN parasites, appelées transposons, qui favorisent leur propre propagation et leur propre transmission, plutôt que celles de leur hôte.. Leurs mouvements (transposition) au sein et entre les génomes ont de profondes conséquences sur le code génétique – entraînant parfois des maladies, mais aussi conduisant à la diversité génétique et à l'évolution. Les protéines de transposase induisent ce mouvement en exécutant toutes les réactions chimiques requises.
De manière frappante, ces protéines ont été réutilisées à plusieurs reprises au cours de l'évolution pour produire de nouvelles fonctions biologiques bénéfiques pour l'hôte. Un excellent exemple est le système immunitaire des vertébrés, dans lequel les protéines RAG de type transposase aident à assembler de nouveaux gènes à partir de trois pools de parties d'ADN interchangeables (appelés segments de gènes V, D et J). Ce processus s'appelle recombinaison V (D) J et équipe les cellules immunitaires d'un ensemble divers de capteurs capables de reconnaître de nombreuses menaces.. , Liu et al. signaler une série de structures d'une transposase qui est un ancêtre de RAG, éclairant l’histoire évolutive de ces protéines.
Les transposases doivent reconnaître plusieurs sites d’ADN, puis les couper et les joindre dans le bon ordre.. Pour comprendre ce processus en plusieurs étapes, nous devons visualiser les structures de la machinerie moléculaire impliquée à toutes les étapes, ce qui représente un défi technique majeur. Liu et al. Nous avons maintenant fait appel à une puissante combinaison de deux techniques – la cristallographie aux rayons X et la microscopie cryogénique à électrons à une seule particule – pour décrire plusieurs étapes de la transposition avec un niveau de détail remarquable, fournissant ainsi un «film» moléculaire du processus.
Les réalisations des auteurs s’appuient sur de nombreuses années d’études structurelles sur les transposases– et RAG–, offrant une vision de plus en plus complète de leurs fonctions et aidant à relier les points entre les réarrangements «égoïstes» de l'ADN des transposases et les fonctions essentielles qui en ont découlé. Les transposases sont maintenant connues pour avoir une unité centrale catalytique et diverses parties supplémentaires qui se lient à l'ADN ou contrôlent la fonction de la transposase. Ils agissent généralement par paires, chaque dimère contenant deux segments de l'ADN du transposon.
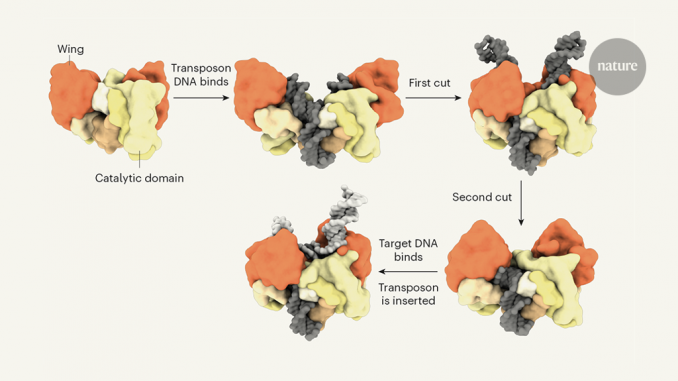
La transposase étudiée par Liu et al. intervient dans le mouvement d’un transposon appelé Transib et a été isolé d’un papillon de nuit (Helicoverpa zea). De manière appropriée, la forme du complexe de transposase ressemble à celle d’un papillon de nuit: chaque "aile" provient d’une des deux molécules de transposase du complexe et est formée principalement d’une région protéique appelée domaine de liaison de zinc (ZnB). Les ailes fournissent de nombreuses interactions avec l’ADN, qui forme les ‘antennes’ (Fig. 1).
Comme beaucoup de transposons, Transib se déplace par un procédé couper-coller: sa transposase la sépare du génome en utilisant un noyau catalytique présent dans de nombreuses transposases et l'insère ailleurs dans le génome.. Les structures de Liu et de ses collaborateurs en cinq étapes de la transposition de Transib révèlent maintenant les changements de conformation remarquables de la protéine au cours de ce processus.
Les auteurs constatent peut-être plus particulièrement que les ailes de ZnB sont constamment en mouvement: elles se déroulent lorsque l'ADN du transposon arrive pour la première fois, puis se referment et s'ouvrent à nouveau pendant le reste du processus. Ce «battement» accompagne d’impressionnantes acrobaties d’ADN, qui introduisent différentes parties de l’ADN dans le noyau de la protéine pour un clivage et une jonction ordonnés. Remarquablement, les domaines ZnB aident à capturer non seulement le transposon, mais également les molécules cibles dans lesquelles Transib sera inséré – s'ouvrant d'abord pour laisser de la place aux molécules, puis se fermant pour bloquer la région de l'ADN cible dans laquelle Transib sera intégré dans le noyau. Les structures montrent également que la partie terminale de la protéine (la queue carboxy-terminale; CTT) contient trois courtes hélices a qui forment une structure en accordéon, qui relie les ailes en mouvement au «corps» du complexe. Structures de transposase rapportées précédemment– ont révélé des caractéristiques globales similaires, mais les mouvements de la transposase Transib sont beaucoup plus étendus.
Les complexes protéiques RAG consistent en deux protéines RAG1 de type transposase et deux protéines RAG2. Dans ces complexes, ZnB est présent dans RAG1, mais est plus fixé que dans la transposase Transib; et aucune partie n'est présente c'est analogue à CTT. RAG2, essentiel à la recombinaison de V (D) J mais absent de la plupart des transposases, est situé au-dessus des ailes de RAG1 et contient une grande partie de l'ADN. – autant que le ZnB et le CTT le font dans la transposase Transib.
Contrairement aux transposases, le complexe RAG coupe et supprime les séquences d'ADN distinctes trouvées entre les segments des gènes V, D et J, en coordonnant étroitement le processus pour s'assurer que différents types de segments sont ensuite connectés. Dans les cellules, RAG empêche également la réinsertion de l'ADN retiré ailleurs dans le génome, afin d'éviter des modifications potentiellement néfastes du code génétique. Mais comment ces fonctions ont-elles évolué? Une proposition logique implique RAG2, mais une étude récente de ProtoRAG – un parent de RAG trouvé chez les invertébrés qui contient RAG2 mais agit toujours comme une transposase – montre que les choses sont plus compliquées. Les éléments de RAG1 et de RAG2 aident à coordonner le clivage de l'ADN et à empêcher l'insertion.
Les conclusions de Liu et ses collègues jettent une lumière nouvelle sur le rôle du RAG2, montrant qu’il remplit de nombreuses fonctions du ZnB, tout en augmentant la rigidité de l’ensemble du complexe RAG par rapport à celle du complexe de la transposase. Il lie l'ADN aux segments V, D et J plus étroitement que le ZnB ne se lie au transposon, et ne subit pas de tels changements conformationnels (qui peuvent nécessiter beaucoup d'énergie, et donc réduire l'efficacité). Cette rigidité accrue et cette liaison étroite pourraient contribuer à assurer la coordination moléculaire stricte requise pour la recombinaison V (D) J. Cela pourrait également empêcher la libération de segments d'ADN clivés et / ou empêcher la réouverture des ailes pour accepter toute autre molécule d'ADN, empêchant ainsi la réinsertion de l'ADN retiré ailleurs dans le génome. Si les ailes ne s'ouvrent pas, tout ADN entrant devrait se plier à un angle d'environ 150 ° avant d'entrer dans la protéine, ce qui n'est pas facile à faire.
Notez que Liu et al. n'ont pas été en mesure d'observer directement la structure de la transposase en complexe avec l'ADN cible intact. Il reste donc à voir si l'ADN cible se lie d'abord à la transposase sous une forme relâchée, puis est forcé dans une courbure sévère à 150 °. Les auteurs n'ont pas non plus observé de complexe dans lequel la transposase se lie à l'ADN de transposon intact, de sorte que le noyau catalytique soit suffisamment proche des extrémités pour les cliver. au lieu de cela, les auteurs ont observé un ADN de transposon intact lié à ses extrémités éloignées du centre catalytique. Dans RAG, une grande torsion dans l'ADN est nécessaire pour positionner ses points de rupture avec précision pour les coupes. Une torsion similaire pourrait se produire dans Transib, mais d’autres explications sont également possibles.
Des efforts sont maintenant nécessaires pour définir les fonctions exactes de RAG2. Curieusement, le complexe RAG sans cellules peut facilement insérer l’ADN excisé dans une autre molécule d’ADN., (un ADN cible). Des structures de RAG avec un ADN cible lié doivent donc être obtenues – idéalement, à la fois avec la cible intacte et après insertion. Ces structures montreront si l'ADN cible devient aussi fortement courbé que dans la transposase Transib et montreront comment RAG2 affecte la liaison de l'ADN cible et son insertion de l'ADN excisé.
D'autres protéines pourraient être nécessaires pour promouvoir la fonction de RAG. Cette possibilité a déjà été étudiée, mais la disponibilité de nouvelles structures et méthodes offre de nouvelles possibilités de recherche. Par exemple, les grands assemblages moléculaires peuvent maintenant être étudiés à l'intérieur de cellules à l'aide d'une technique appelée tomographie électronique., et les interactions moléculaires peuvent être sondées avec des méthodes avancées de spectrométrie de masse. L'analyse des données génomiques de différentes espèces sera également utile pour identifier les ancêtres des protéines RAG autres que ProtoRAG et la transposase Transib, et pour explorer ainsi leur histoire évolutive. Ces recherches aideront à expliquer comment les éléments génétiques parasitaires peuvent être réutilisés pour des fonctions biologiques cruciales.
[ad_2]