[ad_1]
Au cours de la dernière décennie, la diminution des coûts de synthèse chimique de l'ADN et l'amélioration des méthodes d'assemblage de fragments d'ADN ont permis aux chercheurs d'étendre la biologie synthétique au niveau de la génération de chromosomes et de génomes entiers. Jusqu'à présent, l'ADN synthétique a été construit avec un million de paires de bases, notamment un ensemble de chromosomes de la levure. Saccharomyces cerevisiae et plusieurs versions du génome de la bactérie Mycoplasma Mycoides,. Maintenant, Fredens et al. signaler l'achèvement d'une version synthétique de 4 millions de paires de base de la Escherichia coli génome. C’est un point de repère dans le domaine émergent de la génomique synthétique et elle applique enfin cette technologie à la bactérie la plus performante du laboratoire.
La génomique synthétique offre une nouvelle façon de comprendre les règles de la vie, tout en faisant évoluer la biologie synthétique vers un avenir dans lequel les génomes peuvent être écrits pour la conception. Les pionniers du domaine – les chercheurs de l'Institut J. Craig Venter de Rockville (Maryland) – ont utilisé cette méthode pour mieux définir l'ensemble minimal de gènes requis pour une cellule libre. En adoptant une approche qui consiste à redéfinir les segments du génome par ordinateur, à synthétiser chimiquement les fragments puis à les assembler, ces pionniers ont réussi en réduisant la taille de la M. mycoides génome d'environ 50%. Faire la même chose avec seulement des outils d’édition du génome serait beaucoup plus laborieux, comme le travail passé avec E. coli démontre: ici, les méthodes de délétion de gènes ont éliminé, au mieux, seulement 15% du génome.
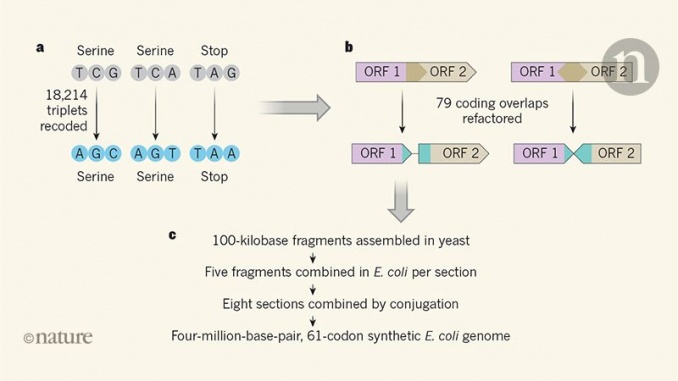
Fredens et ses collègues ont utilisé ce génome réduit de E. coli en tant que modèle pour un génome synthétique avec un autre type de minimisation à l'esprit – la réduction de codon. Le code génétique a une redondance inhérente: il existe 64 codons (triplets de «lettres» ou bases) pour coder seulement 20 acides aminés plus les points «début» et «arrêt» qui marquent le début et la fin d'un tronçon de codage de protéines. séquence. Cette redondance signifie, par exemple, qu'il existe six codons codant pour la sérine d'acides aminés et trois codons d'arrêt possibles. À travers la conception, la synthèse et l’assemblage, Fredens et al. ont pu construire un E. coli génome qui utilise seulement 61 des 64 codons disponibles dans ses séquences codant pour les protéines, en remplaçant deux codons sérine et un codon stop par des synonymes (les codons sont "orthographiés" différemment mais donnent la même instruction). Des travaux antérieurs utilisant des outils d’édition du génome ont déjà produit une synthèse E. coli qui utilise seulement 63 des 64 codons, mais cela nécessitait que les codons d'arrêt avec la séquence TAG (il n'y en avait que 321 autour du génome) soient remplacés par un autre codon d'arrêt. La réduction à 61 codons a nécessité la modification de 18 214 codons, nécessitant une approche de synthèse du génome.
Fredens et ses collègues ont construit leur synthétique E. coli génome en utilisant des méthodes d'assemblage d'ADN à grande échelle et d'intégration du génome qu'ils avaient développées précédemment pour sonder les limites des changements de codons dans E. coli. Dans leur approche (Fig. 1), l’ADN est conçu par ordinateur, synthétisé chimiquement et assemblé en fragments de 100 kilobases dans des vecteurs S. cerevisiae; ces vecteurs sont ensuite repris par E. coli et intégré dans le génome à la place directe de la région naturelle équivalente. En itérant cinq fois ce processus, des sections d'ADN de 500 kilobases ont été remplacées par des versions synthétiques. Huit souches de E. coli ont été produits de cette manière, chacun abritant des sections d’ADN synthétique qui couvraient une région différente du génome. Ces sections ont ensuite été combinées en utilisant des méthodes de conjugaison pour former le génome synthétique complet.
La construction à grande échelle a connu un succès impressionnant, avec des taux de mutation très faibles hors cible, mais ne s'est pas déroulée sans difficultés. Beaucoup de gènes dans le E. coli le génome se chevauchent partiellement avec d’autres, et dans 91 cas, les régions qui se chevauchaient contenaient des codons qu’il fallait changer. Ceci est complexe car des modifications synonymes dans une séquence codant une protéine peuvent altérer les acides aminés codés par celle qui se chevauche. Pour s’y attaquer, l’équipe a «refactorisé» 79 sites dans le génome, en dupliquant la séquence afin de séparer les séquences de codage superposées en séquences codées individuelles (Fig. 1). Bien que cette approche ait généralement été couronnée de succès, elle a nécessité un débogage prudent dans quelques cas où le refactoring a également modifié la régulation des gènes.
La souche finale s’est révélée viable et a pu croître dans diverses conditions de laboratoire, bien qu’elle soit un peu moins vigoureuse que sa contrepartie naturelle. Il n’utilise plus le codon d’arrêt TAG ni les deux codons de sérine TCG et TCA, de sorte que la machinerie cellulaire qui les reconnaît peut maintenant être supprimée ou réaffectée pour recruter des acides aminés «non canoniques» au-delà des 20 généralement utilisés par la plupart des cellules vivantes. Ce recrutement s’est déjà révélé utile dans le codon à 63 codons. E. coli, à la fois pour des projets de biotechnologie, dans lesquels des acides aminés non canoniques sont codés dans les positions de séquence souhaitées pour fournir des résidus pouvant participer à des réactions chimiques que les protéines naturelles ne peuvent pas; et pour des raisons de biosécurité, en ce que le transfert naturel des informations lisibles codées par l'ADN à l'intérieur et à l'extérieur du E. coli est limité car la cellule fonctionne avec un code génétique légèrement différent du reste du monde naturel. Attendez-vous à ce que toutes ces applications soient étendues dans le nouveau codon 61 E. coli, qui pourrait coder l’utilisation de plus d’un acide aminé non canonique et générer un pare-feu génétique plus strict (car 3 des 64 codons ne sont plus reconnus).
La synthèse d'un génome de 4 millions de paires de bases et la réduction du code génétique à 61 codons sont de nouveaux enregistrements pour la génomique de synthèse, mais pourraient ne pas durer beaucoup plus longtemps. Le consortium international Sc2.0 se rapproche de la synthèse des 16 chromosomes de la paire de 12 millions de bases S. cerevisiae génome – le premier génome de synthèse d'un organisme eucaryote, le groupe qui comprend les plantes, les animaux et les champignons – et la synthèse d'un codon 57 E. coli le génome est également en cours,. Un génome de la bactérie Salmonella Typhimurium qui a deux codons en moins que l'organisme naturel est également en cours de construction. Cela pourrait un jour permettre à des bactéries possédant un génome synthétique d'être utilisées en tant que technologies à base de cellules dans l'intestin humain.
D'un point de vue technologique, l'aspect le plus intéressant de tous ces projets est que les processus de construction du génome synthétique sont remarquablement similaires, des sections en kilobases d'ADN synthétisé étant assemblées (par le processus de recombinaison homologue) en 50 à 100 kilobases. des morceaux dans des cellules de levure, ces morceaux étant ensuite utilisés pour remplacer des séquences naturelles à l'intérieur de l'organisme cible (par des méthodes de recombinaison sélectionnables). La standardisation des méthodes permettra d'automatiser les étapes et de permettre à davantage de groupes de recherche d'entrer sur le terrain. La minimisation des génomes et la réduction des codons ne sont que les premières utilisations de cette nouvelle technologie, qui pourrait un jour nous donner des génomes fonctionnellement réorganisés et des génomes conçus sur mesure pour diriger les cellules vers des tâches spécialisées.
Inscrivez-vous pour le quotidien Briefing Nature Courriel
Restez au courant de ce qui compte dans la science et pourquoi, choisi à la main La nature et d'autres publications dans le monde.
S'inscrire
[ad_2]