[ad_1]
Adelman, K. & Lis, J. T. Pause proximale du promoteur de l'ARN polymérase II: rôles émergents chez les métazoaires. Nat. Rev. Genet. 13, 720–731 (2012).
Harlen, K.M. & Churchman, L.S. Le code et au-delà: régulation de la transcription par le domaine carboxy-terminal de l'ARN polymérase II. Nat. Rev. Mol. Cellule biol. 18263–273 (2017).
Levine, M., Cattoglio, C. & Tjian, R. Looping: un retour en avant: la transcription entre dans une nouvelle ère. Cellule 157, 13-25 (2014).
Sainsbury, S., Bernecky, C. et Cramer, P. Bases structurelles de l'initiation de la transcription par l'ARN polymérase II. Nat. Rev. Mol. Cellule biol. 16129–143 (2015).
Eick, D. & Geyer, M. Le code de domaine carboxy-terminal (CTD) de l'ARN polymérase II. Chem. Tour. 1138456–8490 (2013).
Ebmeier, C.C. et al. La kinase CDK7 de TFIIH humaine régule les modifications de la chromatine associées à la transcription. Rapports de cellule 20, 1173-1186 (2017).
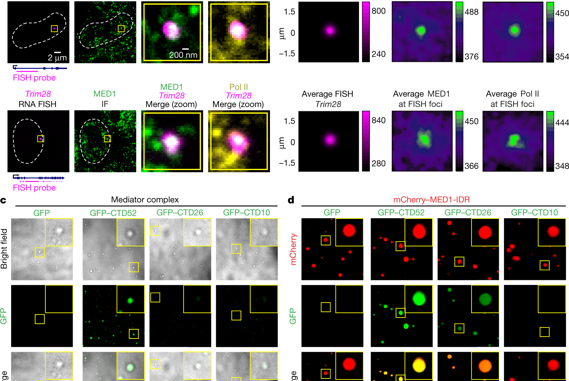
Cho, W. K. et al. Des amas de médiateurs et d'ARN polymérase II s'associent dans des condensats dépendants de la transcription. Science 361412–415 (2018).
Sabari, B.R. et al. La condensation de coactivateur au niveau des super-amplificateurs relie la séparation de phase et le contrôle des gènes. Science 361eaar3958 (2018).
Spector, D. L. & Lamond, A. I. Mouchetures nucléaires. Printemps froid Harb. Perspective. Biol. 3, a000646 (2011).
Chen, Y. et al. Cartographie de l'organisation du génome 3D par rapport aux compartiments nucléaires en utilisant TSA-seq comme règle cytologique. J. Cell Biol. 217, 4025 à 4048 (2018).
Quinodoz, S.A. et al. Les concentrateurs interchromosomiques d'ordre supérieur modèlent l'organisation du génome 3D dans le noyau. Cellule 174744–757 (2018).
Hall, L. L., Smith, K. P., Byron, M. et Lawrence, J. B. Anatomie moléculaire d'un chatoiement. Anat. Rec. Une discov. Mol. Cellule. Evol. Biol. 288A664–675 (2006).
Allen, B. L. & Taatjes, D. J. Le complexe Mediator: un intégrateur central de la transcription. Nat. Rev. Mol. Cellule biol. 16155–166 (2015).
Boija, A. et al. Les facteurs de transcription activent les gènes grâce à la capacité de séparation de phase de leurs domaines d'activation. Cellule 175, 1842-1855 (2018).
Zhang, X. et al. MED1 / TRAP220 existe principalement dans une sous-population TRAP / Mediator enrichie en ARN polymérase II et est nécessaire pour la transcription médiée par ER. Mol. Cellule 19, 89–100 (2005).
Banani, S. F., Lee, H. O., Hyman, A. A. & Rosen, M. K. Condensats biomoléculaires: organisateurs de la biochimie cellulaire. Nat. Rev. Mol. Cellule biol. 18285-298 (2017).
Hnisz, D., K. Shrinivas, R. A. Young, Chakraborty, A. K. et Sharp, A. Un modèle de séparation de phases pour le contrôle de la transcription. Cellule 169, 13–23 (2017).
Shin, Y. & Brangwynne, C. P. Condensation en phase liquide dans la physiologie cellulaire et la maladie. Science 357, ea4382 (2017).
Hnisz, D. et al. Super activateurs dans le contrôle de l'identité cellulaire et de la maladie. Cellule 155, 934 à 947 (2013).
Braunschweig, U., Gueroussov, S., A. Plocik, A.M., Graveley, B.R. et Blencowe, B.J., Intégration dynamique de l'épissage dans les voies de régulation des gènes. Cellule 1521252-1269 (2013).
Herzel, L., Ottoz, D.S., Alpert, T. & Neugebauer, K.M. Base tactile d'épissage et de transcription: assemblage et fonction de la transcription dans le spliceosome. Nat. Rev. Mol. Cellule biol. 18637–650 (2017).
Hsin, J. P. et Manley, J. L. L'ARN polymérase II CTD coordonne la transcription et le traitement de l'ARN. Genes Dev. 262119-2137 (2012).
Kotake, Y. et al. Le facteur d’épissage SF3b comme cible du produit naturel antitumoral, le pladiénolide. Nat. Chem. Biol. 3570-575 (2007).
Akhtar, M.S. et al. La kinase TFIIH place des marques bivalentes sur le domaine carboxy-terminal de l'ARN polymérase II. Mol. Cellule 34, 387–393 (2009).
Czudnochowski, N., Bösken, C. A. et Geyer, M. La sérine-7, mais pas la phosphorylation de la sérine-5, amorce l'ARN polymérase II CTD pour la reconnaissance de P-TEFb. Nat. Commun. 3, 842 (2012).
Kwon, I. et al. Liaison régulée par la phosphorylation de l'ARN polymérase II à des polymères fibreux de domaines de faible complexité. Cellule 1551049-1060 (2013).
Long, J. C. & Caceres, J. F. La famille de protéines SR des facteurs d'épissage: régulateurs maîtres de l'expression des gènes. Biochem. J. 417, 15-27 (2009).
McCracken, S. et al. Le domaine C-terminal de l'ARN polymérase II couple le traitement de l'ARNm à la transcription. La nature 385357-361 (1997).
Boehning, M. et al. Regroupement de l'ARN polymérase II par séparation de phase dans les domaines carboxy-terminaux. Nat. Struct. Mol. Biol. 25833 à 840 (2018).
Lu, H. et al. Mécanisme de séparation de phases pour l'hyperphosphorylation C-terminale de l'ARN polymérase II. La nature 558318–323 (2018).
Bolte, S. & Cordelières, F. P. Visite guidée dans l'analyse de la colocalisation subcellulaire en microscopie optique. J. Microsc. 224213-232 (2006).
Meyer, K.D. et al. L'activité coopérative de cdk8 et de GCN5L au sein de Mediator dirige la phosphoacétylation en tandem de l'histone H3. EMBO J. 271447-1457 (2008).
Shen, L., Shao, N., Liu, X. et Nestler, E. ngs.plot: extraction et visualisation rapides des données de séquençage de la prochaine génération grâce à l'intégration de bases de données génomiques. BMC Génomique 15284 (2014).
Grimm, J.B. et al. Méthode générale d'amélioration des fluorophores pour la microscopie à cellules vivantes et à molécule unique. Nat. Les méthodes 12, 244–250 (2015).
Lambert, T. et Shao, L. tlambert03 / LLSpy v.0.3.8. (2018).
Sergé, A., Bertaux, N., Rigneault, H. et Marguet, D. Traçage dynamique à cibles multiples pour sonder la cartographie spatio-temporelle des membranes cellulaires. Nat. Les méthodes 5687–694 (2008).
Verdaasdonk, J. S., Lawrimore, J. et Bloom, K. Détermination du nombre absolu de protéines par microscopie quantitative à fluorescence. Méthodes Cell Biol. 123, 347–365 (2014).
Younis, I. et al. Les écrans rapporteurs d'épissage à réponse rapide identifient les régulateurs différentiels de l'épissage constitutif et alternatif. Mol. Cellule. Biol. 301718-1728 (2010).
Banani, S. F. et al. Contrôle de la composition des corps cellulaires séparés par des phases. Cellule 166, 651–663 (2016).
Wang, J. et al. Une grammaire moléculaire régissant les forces motrices de la séparation de phase des protéines de liaison à l'ARN de type prion. Cellule 174688–699 (2018).
[ad_2]