[ad_1]
Eckburg, P. B. et al. Diversité de la flore microbienne intestinale humaine. Science 3081635-1638 (2005).
Gill, S. R. et al. Analyse métagénomique du microbiome intestinal humain. Science 312, 1355-1359 (2006).
Costello, E.K. et al. Variation de la communauté bactérienne dans les habitats du corps humain à travers l'espace et le temps. Science 3261694-1697 (2009).
Hsiao, E. Y. et al. Les microbiotes modulent les anomalies comportementales et physiologiques associées aux troubles du développement neurologique. Cellule 1551451–1463 (2013).
Gopalakrishnan, V. et al. Le microbiome intestinal module la réponse à l'immunothérapie anti-PD-1 chez les patients atteints de mélanome. Science 35997-103 (2018).
Matson, V. et al. Le microbiome commensal est associé à l'efficacité anti-PD-1 chez les patients atteints de mélanome métastatique. Science 359104-108 (2018).
Routy, B. et al. Le microbiome intestinal influe sur l'efficacité de l'immunothérapie à base de PD-1 contre les tumeurs épithéliales. Science 35991–97 (2018).
Haiser, H.J. et al. Prédiction et manipulation de l'inactivation cardiaque de médicaments par la bactérie intestinale humaine Eggerthella lenta. Science 341295–298 (2013).
Taur, Y. et al. Les effets de la diversité bactérienne du tractus intestinal sur la mortalité après une greffe allogénique de cellules souches hématopoïétiques. Du sang 1241174-1182 (2014).
Schloissnig, S. et al. Paysage de la variation génomique du microbiome intestinal humain. La nature 493, 45–50 (2013).
Franzosa, E.A. et al. Identifier des microbiomes personnels à l'aide de codes métagénomiques. Proc. Natl Acad. Sci. Etats-Unis 112E2930 à E2938 (2015).
Dominguez-Bello, M.G. et al. Le mode d'administration détermine l'acquisition et la structure du microbiote initial dans plusieurs habitats corporels chez le nouveau-né. Proc. Natl Acad. Sci. Etats-Unis 10711971-11975 (2010).
Koenig, J.E. et al. Succession de consortiums microbiens dans le microbiome intestinal en développement du nourrisson. Proc. Natl Acad. Sci. Etats-Unis 1084578–4585 (2010).
Vatanen, T. et al. Le microbiome intestinal humain dans le diabète de type 1 précoce de l'étude TEDDY. La nature 562589-594 (2018).
Yatsunenko, T. et al. Microbiome intestinal humain vu à travers l’âge et la géographie. La nature 486222-222 (2012).
Pasolli, E. et al. Vaste diversité de microbiomes humains inexplorée révélée par plus de 150 000 génomes provenant de métagénomes couvrant l’âge, la géographie et le mode de vie. Cellule 176, 649–662.e620 (2019).
Faith, J. J. et al. La stabilité à long terme du microbiote intestinal humain. Science 3411237439 (2013).
Gajer, P. et al. Dynamique temporelle du microbiote vaginal humain. Sci. Trad. Med. 4, 132ra152 (2012).
Gevers, D. et al. Le projet du microbiome humain: une ressource communautaire pour le microbiome humain en santé. PLoS Biol. dix, e1001377 (2012).
Turnbaugh, P. J. et al. Le projet de microbiome humain. La nature 449804–810 (2007).
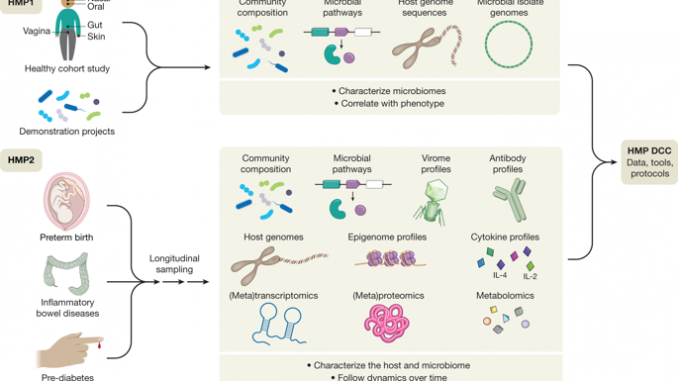
Consortium du projet sur le microbiome humain. Structure, fonction et diversité du microbiome humain sain. La nature 486, 207-214 (2012). Étude HMP1 analysant 5 177 profils de séquençage des gènes de l'ARNr 16S et 681 métagénomes de fusil de chasse couvrant jusqu'à 18 sites corporels et trois points de temps chacun chez 242 adultes en bonne santé; gamme de base établie de diversité taxonomique à l'intérieur et entre les sites corporels, ainsi que personnalisation des souches et points communs fonctionnels.
Consortium du projet sur le microbiome humain. Un cadre pour la recherche sur le microbiome humain. La nature 486215-221 (2012).
Lloyd-Price, J. et al. Souches, fonctions et dynamiques dans le projet de microbiome humain élargi. La nature 550, 61–66 (2017). Deuxième vague de données HMP1 (HMP1-II) étendant l'analyse et la ressource de données à 2 355 métagénomes totaux de fusil de chasse chez 265 adultes en bonne santé; personnalisation quantifiée de la souche et dynamique de rétention dans le temps, ainsi que l'identification de fonctions de communauté microbienne spécifiques à un créneau et associées à l'hôte.
Ravel, J. et al. Microbiome vaginal de femmes en âge de procréer. Proc. Natl Acad. Sci. Etats-Unis 1084680–4687 (2011).
Fettweis, J. M. et al. Différences dans le microbiome vaginal chez les femmes afro-américaines par rapport aux femmes d'ascendance européenne. Microbiologie 160, 2272 à 2282 (2014).
Kong H. H. et al. Les changements temporels dans le microbiome cutané associés aux poussées de maladie et au traitement chez les enfants atteints de dermatite atopique. Génome res. 22, 850–859 (2012).
Alekseyenko, A. V. et al. Différenciation communautaire du microbiote cutané dans le psoriasis. Microbiome 131 (2013).
Wylie, K.M., Mihindukulasuriya, K.A., Sodergren, E., Weinstock, G.M. et Storch, G.A. Analyse de séquence du virome humain chez des enfants fébriles et afébriles. PLoS One 7, e27735 (2012).
Lewis, J.D. et al. L’inflammation, les antibiotiques et le régime alimentaire en tant que facteurs de stress environnementaux du microbiome intestinal dans la maladie de Crohn de l’enfant. Microbe hôte cellulaire 18, 489–500 (2015).
Zupancic, M. L. et al. Analyse du microbiote intestinal dans l'ordre Amish ancien et sa relation avec le syndrome métabolique. PLoS One 7, e43052 (2012).
Erickson, A. R. et al. La métagénomique / métaprotéomique intégrée révèle les signatures de la maladie de Crohn chez l’hôte-microbiote humain. PLoS One 7, e49138 (2012).
Frank, D. N. et al. Le phénotype et le génotype de la maladie sont associés à des modifications du microbiote intestinal associé aux maladies inflammatoires de l'intestin. Inflamm. Intestin Dis. 17179–184 (2011).
La Rosa, P. S. et al. Progression structurée des populations bactériennes dans l'intestin du prématuré. Proc. Natl Acad. Sci. Etats-Unis 111, 12522 à 12527 (2014).
Fodor, A. A. et al. Les taxons «les plus recherchés» du microbiome humain pour le séquençage du génome entier. PLoS One 7, e41294 (2012).
Nelson, K. E. et al. Un catalogue de génomes de référence du microbiome humain. Science 328994–999 (2010).
Wylie, K.M. et al. Nouveaux taxons bactériens dans le microbiome humain. PLoS One 7, e35294 (2012).
Li, K., Bihan, M., Yooseph, S. et Methé, B. A. Analyses de la diversité microbienne dans le microbiome humain. PLoS One 7, e32118 (2012).
Aagaard, K. et al. Stratégie du projet sur le microbiome humain pour un échantillonnage complet du microbiome humain et son importance. FASEB J. 27, 1012-1022 (2013).
Schloss, P. D., Gevers, D. et Westcott, S. L. Réduction des effets de l'amplification par PCR et du séquençage d'artefacts sur des études basées sur l'ARNr 16S. PLoS One 6, e27310 (2011).
Groupe de travail sur la génération de données du projet Microbiome humain du Consortium Jumpstart. Évaluation du profilage communautaire fondé sur l'ADNr 16S pour la recherche sur le microbiome humain. PLoS One 7, e39315 (2012).
Gevers, D., Pop, M., Schloss, P. D. et Huttenhower, C. Bioinformatics pour le projet du microbiome humain. PLoS Comput. Biol. 8e1002779 (2012).
Huse, S. M., Ye, Y., Zhou, Y. et Fodor, A. A. Un microbiome humain de base tel que vu à travers des grappes de séquences d'ARNr 16S. PLoS One 7, e34242 (2012).
Markowitz, V.M. et al. IMG / M-HMP: un système d’analyse comparative de métagénomes pour le projet du microbiome humain. PLoS One 7, e40151 (2012).
Cantarel, B.L., Lombard, V. & Henrissat, B. Utilisation complexe des glucides par le microbiome humain sain. PLoS One 7, e28742 (2012).
Rho, M., Wu, Y. W., Tang, H., Doak, T. G. et Ye, Y. Diverses CRISPR évoluant dans les microbiomes humains. PLoS Genet. 8, e1002441 (2012).
Faust, K. et al. Relations de cooccurrence microbienne dans le microbiome humain. PLoS Comput. Biol. 8, e1002606 (2012).
Abubucker, S. et al. Reconstruction métabolique des données métagénomiques et son application au microbiome humain. PLoS Comput. Biol. 8e1002358 (2012).
Consortium du réseau de recherche intégrative HMP (iHMP). Le projet intégratif de microbiome humain: analyse dynamique des profils omiques microbiome-hôte au cours de périodes de maladie et de santé humaines. Microbe hôte cellulaire 16, 276 à 289 (2014).
Lloyd-Price, J. et al. Multi-omiques de l'écosystème microbien intestinal dans les maladies inflammatoires de l'intestin. La nature (2019). HMP2 Étude de la base de données Multi’omics sur les maladies intestinales inflammatoires (IBDMDB) portant sur 2 965 échantillons de selles, de biopsie et de sang prélevés chez 132 personnes atteintes de la maladie de Crohn, de la colite ulcéreuse ou de non-contrôle (sujets témoins) suivies longitudinalement pendant un an chacune; profil multi-omique identifiant des taxons, des fonctions exprimées, des pools de métabolites et l'expression génique de l'hôte qui ont été perturbés dans l'intestin lors d'une activité accrue de la maladie, ainsi que des effets systémiques tels que les niveaux d'anticorps sériques.
Fettweis, J. M. et al. Le microbiome vaginal et la naissance prématurée. Nat. Med. (2019). Étude sur la prématurité selon HMP2 portant sur 1 527 grossesses et portant sur 45 naissances spontanées prématurées et 90 témoins appariés, ce qui a permis d'obtenir plus de 12 000 échantillons à profils multiples; Les profils des gènes d’ARNr 16S, métagénomiques, métatranscriptomiques et cytokines ont permis Lactobacillus crispatus, BVAB1, Sneathia amnii, TM7-H1 et autres taxons associés aux accouchements prématurés.
Zhou, W. et al. Multi-omics longitudinaux de la dynamique hôte-microbe dans le prédiabète. La nature (2019). Étude sur le prédiabète HMP2 portant sur 1 092 visites chez 106 participants, décrivant les transcriptomes, les métabolomes, les cytokines et les protéomes de l'hôte, ainsi que les modifications taxonomiques et génomiques microbiennes au cours de périodes saines, les infections virales respiratoires, la vaccination et d'autres perturbations; la plupart des profils moléculaires étaient personnalisés, perturbés lors d'infections et d'immunisations et associés à des modifications de la sensibilité à l'insuline.
Behrman, R. E. & Butler A. S. (éd.) Naissance prématurée: causes, conséquences et prévention (National Academies Press, 2007).
York, T.P., Eaves, L.J., Neale, M.C. et Strauss, J.F. III. La contribution des facteurs génétiques et environnementaux à la durée de la grossesse. Un m. J. Obstet. Gynecol. 210398–405 (2014).
Liu, L. et al. Causes mondiales, régionales et nationales de la mortalité des moins de 5 ans en 2000-2015: une analyse systématique actualisée ayant des implications pour les objectifs de développement durable. Lancette 3883027-3035 (2016).
Mor, G., Aldo, P. et Alvero, A. B. Les aspects immunologiques et microbiens uniques de la grossesse. Nat. Rev. Immunol. 17469–482 (2017).
Romero, R., Dey, S. K. et Fisher, S. J. Travail prématuré: un syndrome, plusieurs causes. Science 345760–765 (2014).
Jefferson, K. K. L'étiologie bactérienne de la naissance prématurée. Adv. Appl. Microbiol. 80, 1–22 (2012).
Serrano, M.G. et al. Diversité racioéthnique dans la dynamique du microbiome vaginal pendant la grossesse. Nat. Med. (2019). Étude HMP2 sur la naissance à terme portant sur 613 femmes enceintes, 1 969 femmes non enceintes et 90 femmes enceintes supplémentaires, suivies d'un profilage longitudinal d'échantillons vaginaux, buccaux, sanguins, urinaires et rectaux; se déplace vers Lactobacilles
communautés dominées avaient tendance à se manifester tôt pendant la grossesse, en particulier chez les femmes d'ascendance africaine ou hispanique.
DiGiulio, D. B. et al. Variation temporelle et spatiale du microbiote humain pendant la grossesse. Proc. Natl Acad. Sci. Etats-Unis 11211060-11065 (2015).
Callahan, B.J. et al. Réplication et affinement d'une signature microbienne vaginale de prématurité dans deux cohortes de femmes américaines racialement distinctes. Proc. Natl Acad. Sci. Etats-Unis 1149966–9971 (2017).
Kindinger, L.M. et al. L'interaction entre le microbiote vaginal, la longueur du col utérin et le traitement vaginal à la progestérone pour le risque d'accouchement prématuré. Microbiome 5, 6 (2017).
Brown, R. G. et al. La dysbiose vaginale augmente le risque de rupture prématurée de la membrane fœtale, de septicémie néonatale et est exacerbée par l’érythromycine. BMC Med. 169 (2018).
Jefferson, K. K. et al. Relation entre le statut en vitamine D et le microbiome vaginal pendant la grossesse. J. Perinatol. (2019).
Zhou, S. S., Tao, Y. H., K. Huang, B. B. et Tao, F. B. Vitamine D et risque d'accouchement prématuré: méta-analyse à jour d'essais contrôlés randomisés et d'études d'observation. J. Obstet. Gynaecol. Res. 43, 247-256 (2017).
Huang, H. et al. Cartographie fine des locus inflammatoires de l'intestin avec résolution à un seul variant. La nature 547173–178 (2017).
Manichanh, C. et al. Réduction de la diversité du microbiote fécal dans la maladie de Crohn révélée par une approche métagénomique. Intestin 55, 205-211 (2005).
Frank, D. N. et al. Caractérisation moléculaire-phylogénétique des déséquilibres de la communauté microbienne dans les maladies inflammatoires de l'intestin chez l'homme. Proc. Natl Acad. Sci. Etats-Unis 10413780-13785 (2007).
Kugathasan, S. et al. Prévision de l'évolution d'une maladie compliquée chez les enfants nouvellement diagnostiqués de la maladie de Crohn: une étude de cohorte multicentrique à l'origine. Lancette 3891710-1718 (2017).
Narula, N. et al. Revue systématique et méta-analyse: transplantation de microbiote fécal pour le traitement de la colite ulcéreuse active. Inflamm. Intestin Dis. 231702–1709 (2017).
Jeon, S. R., Chai, J., Kim, C. et Lee, C. H. Données actuelles sur la prise en charge des maladies inflammatoires de l'intestin par transplantation de microbiote fécal. Curr. Infecter. Dis. Représentant. 2021 (2018).
Morgan, X. C. et al. Dysfonctionnement du microbiome intestinal dans la maladie inflammatoire de l'intestin et son traitement. Génome biol. 13, R79 (2012).
Hall, A. B. et al. Un roman Ruminococcus gnavus clade enrichi chez les patients atteints de maladies inflammatoires de l'intestin. Génome Med. 9103 (2017).
Schirmer, M. et al. Dynamique de la métatranscription dans le microbiome intestinal de la maladie inflammatoire de l'intestin. Nat. Microbiol. 3337–346 (2018).
Franzosa, E.A. et al. Structure du microbiome intestinal et activité métabolique dans les maladies inflammatoires de l'intestin. Nat. Microbiol. 4293-305 (2019).
Mallick, H. et al. Profil métabolomique prédictif des communautés microbiennes à l'aide de séquences d'amplicons ou métagénomiques. Nat. Commun. (dans la presse).
Franzosa, E.A. et al. Profil fonctionnel des métagénomes et des métatranscriptomes au niveau des espèces. Nat. Les méthodes 15962 à 968 (2018).
McIver, L. J. et al. bioBakery: un environnement d’analyse méta’omique. Bioinformatique 34, 1235-1237 (2018).
Willing, B. P. et al. Une étude de pyroséquençage chez des jumeaux montre que les profils microbiens gastro-intestinaux varient en fonction des phénotypes des maladies inflammatoires de l'intestin. Gastroentérologie 139, 1844–1854.e1841 (2010).
Gevers, D. et al. Le microbiome naïf de traitement dans la maladie de Crohn à l'apparition récente. Microbe hôte cellulaire 15, 382–392 (2014).
Cowie, C.C. et al. Comptabilité complète du diabète et du prédiabète dans la population américaine en 1988-1994 et 2005-2006. Traitements diabétiques 32287-294 (2009).
Pickup, J. C. Inflammation et immunité innée activée dans la pathogenèse du diabète de type 2. Traitements diabétiques 27813 à 823 (2004).
Qin, J. et al. Une étude d'association à l'échelle du métagénome du microbiote intestinal dans le diabète de type 2. La nature 490, 55–60 (2012).
Gamble, D. R., Kinsley, M. L., FitzGerald, M. G., Bolton, R. et Taylor, K. W. Anticorps viraux dans le diabète sucré. BMJ 3627-630 (1969).
Mehta, S. H. et al. Prévalence du diabète sucré de type 2 chez les personnes atteintes du virus de l'hépatite C aux États-Unis. Ann. Interne. Med. 133592-599 (2000).
Chen, R. et al. Le profilage personnel en ombilique révèle des phénotypes moléculaires et médicaux dynamiques. Cellule 148, 1293 à 1307 (2012).
Piening, B.D. et al. Profils omiques personnels intégratifs pendant les périodes de prise et de perte de poids. Cell Syst. 6157–170 e158 (2018).
Schüssler-Fiorenza Rose, S. et al. Une approche big data longitudinale pour une santé de précision. Nat. Med. 25792–804 (2019). Profil Omique Personnalisé Intégratif HMP2 de 109 personnes enrichies pour le risque de diabète de type 2 étudié génomiquement, immunologiquement, transcriptomiquement, protéomiquement, métabolomiquement et en utilisant une surveillance portable; de multiples voies moléculaires étaient associées à la physiopathologie métabolique, cardiovasculaire et oncologique, et un sous-ensemble était prédictif de la résistance à l'insuline.
Wilkinson, M.D. et al. Les principes directeurs de FAIR pour la gestion et la gestion des données scientifiques. Sci. Les données 3160018 (2016).
Équipe d’analyse du portefeuille de microbiomes humains des NIH. Revue des dix années d'activités de recherche sur le microbiome humain dans les National Institutes of Health des États-Unis, exercices financiers 2007-2016. Microbiome 731 (2019).
[ad_2]