[ad_1]
Purification de la transhydrogénase des mitochondries ovines
Toutes les procédures décrites ont été effectuées à 4 ° C. Les mitochondries ont été purifiées à partir de tissu cardiaque ovin frais par centrifugation différentielle et conservées à -80 ° C selon la procédure 3 de la réf. 31. Le jour de la purification, les membranes mitochondriales internes ont été isolées comme décrit pour la purification du complexe respiratoire I32. En bref, les mitochondries (10 g) ont été rompues par homogénéisation dans 100 ml d’eau Milli-Q, puis les membranes ont été séparées par centrifugation à 50 000 ° C.g pendant 45 minutes et remis en suspension dans 100 ml de tampon M (HEPES 20 mM, pH 7,4, NaCl 40 mM, EDTA 1 mM, glycérol à 10% v / v, DTT 2 mM, 0,002% de PMSF). Après un autre cycle de remise en suspension et de centrifugation, les membranes ont été remises en suspension dans 50 ml de tampon M. Du maltose de lauryle néopentylglycol (LMNG; 10%) a été ajouté goutte à goutte à cette suspension à 1% et, après agitation pendant 45 minutes, l'échantillon a été centrifugé à 50 000g pendant 45 min. Le surnageant a été filtré avec un filtre de 0,22 um et chargé sur une colonne échangeuse d'anions Q-Sepharose HP de 45 ml équilibrée dans du tampon QA (HEPES 20 mM pH 7,4, NaCl 40 mM, EDTA 2 mM, glycérol à 10%). , 1 mM de DTT, 0,05% de LMNG). Les protéines ont été élues avec un gradient linéaire de 400 ml avec 0 à 17% de tampon Q-B (tampon Q-A avec 1 M de NaCl). Le NNT a été élué sous forme d’un large pic à environ 110 mM de NaCl et les fractions contenant des quantités substantielles de NNT (sur la base du dosage de l’activité et du profil SDS-PAGE) ont été regroupées et dialysées pendant une nuit contre un excès de volume dix fois supérieur du tampon D (MES, pH 20 mM). 5,8, EDTA 2 mM, glycérol à 10% v / v, DTT 1 mM, 0,01% de LMNG). Cela a abaissé le pH de l'échantillon en dessous de 6 et la conductance en dessous de 2 mS cm−1et le rend apte à être chargé sur une colonne échangeuse de cations SP-Sepharose HP de 45 ml équilibrée dans du tampon SP-A (MES 20 mM, pH 5,8, NaCl 10 mM, EDTA 2 mM, glycérol à 10% v / v, DTT 1 mM , 0,025% de LMNG). L'élution avec un gradient linéaire de 100 ml à 0-20% avec le tampon SP-B (SP-A avec NaCl 1 M) a donné un pic d'élution net de NNT relativement pur à environ 35 mM de NaCl (conductance 4 mS cm−1). Les fractions contenant du NNT ont été concentrées à 4 mg ml−1 utilisant un concentrateur de coupure de 100 kDa et stocké à 30% de glycérol sous azote liquide. Le jour de la préparation d'une grille de cryo-EM, un échantillon de NNT a été décongelé sur de la glace et déposé sur une colonne de filtration sur gel Superose 12 10/300 équilibrée dans du tampon GF (HEPES 20 mM, pH 7,4, NaCl 50 mM, EDTA 1 mM). , 0,002% de LMNG) pour éliminer le détergent en excès et les contaminants protéiques restants. Les fractions de NNT les plus pures et les plus concentrées ont été éluées à environ 10,5 ml et ont été concentrées à 5 mg ml.−1 utilisant un filtre de coupure Millipore 100 kDa et utilisé immédiatement pour la préparation de la grille cryo-EM (Extended Data Fig. 2). Dans certaines purifications, 0,002% de LMNG dans du tampon GF ont été remplacés par 0,05% d'octyl maltoside fluoré (FOM), ce qui a donné un NNT monodispersé et hautement actif; cependant, les grilles adaptées à la collecte de données sur la cryo-EM n'ont pas pu être préparées à partir de préparations de FOM, car les protéines ont commencé à s'agréger sur la grille à des concentrations supérieures à 2 mg ml−1. La forme apo de notre préparation était dépourvue de tout nucléotide lié, ce qui est confirmé par l’absence de réaction cyclique sans addition de NADP exogène.+ (données non présentées).
Microscopie électronique
On a ajouté 0,05% de CHAPS à l'échantillon de protéine avant la préparation de la grille pour améliorer la qualité de la glace et la distribution des particules. Des substrats à une concentration de 5 mM ont été ajoutés à la protéine 15 à 30 min avant la préparation de la grille et les échantillons ont été incubés sur de la glace. Un échantillon de protéine (2,7 µl) a été appliqué sur une grille de cuivre Quantifoil 0,6 / 1 fraîchement libérée et étanchéifié pendant 4 à 6 s à l’aide d’une force de buvardage de 25 à 4 ° C et à 100% d’humidité dans un Vitrobot Mark IV (FEI). Les grilles ont été surgelées dans de l'éthane liquide et stockées dans de l'azote liquide jusqu'à la collecte des données. NNT – NADP+ Les grilles ont été imagées à l'aide d'un microscope électronique Titan Krios 300 kV équipé d'une caméra de sommet K2 Gatan filtrée par énergie d'une largeur de fente de 20 eV au Centre Astbury pour la biologie moléculaire structurale de l'Université de Leeds. Les réseaux NNT – NADPH ont été imagés à l'aide d'un microscope électronique Titan Krios à 300 kV équipé d'une caméra au sommet K2 Gatan filtrée par énergie avec une largeur de fente de 20 eV sur la ligne de lumière CM01, ESRF, Grenoble. Les images ont été collectées avec le logiciel d’acquisition EPU dans un mode super-résolution K2 avec un grossissement nominal de 130 000 × et une taille de pixel physique de 1,065 Å pour le NNT – NADP.+ jeu de données et 1,07 Å pour le jeu de données NNT – NADPH. Une dose totale de 72 e– UNE−2 (NNT – NADP+) ou 70 e– UNE−2 (NNT – NADPH) a été fractionné en 40 trames de 250 ms chacune. Les réseaux Apo-NNT ont été imagés à l'aide d'un microscope électronique Titan Krios à 300 kV équipé d'une caméra K3 à filtre d'énergie Gatan avec une largeur de fente de 20 eV à l'Institut des sciences et de la technologie d'Autriche. La taille des pixels physiques était de 0,84 Å et la dose totale de 90 e– UNE−2 a été fractionné en 88 trames de 36 ms chacune.
Traitement d'image
Nous avons recueilli 2 272 films pour le PNDA+ ensemble de données, 1 722 films pour l'ensemble de données NADPH et 786 films pour l'ensemble de données apo-NNT. Le traitement a été effectué dans RELION 2.133 sauf indication contraire. Les images de la vidéo ont été alignées à l'aide de MotionCor234 et les paramètres de la fonction de transfert de contraste initial (CTF) ont été estimés à partir d'images moyennées à l'aide de CTFFIND35. La sélection automatique avec des moyennes de classe 2D comme références a donné 516 395 particules pour le jeu de données NADPH, 1 076 677 particules pour le NADP+ ensemble de données et 500 001 particules pour l’ensemble de données apo. Plusieurs cycles de classification 2D et 3D ont été réalisés pour classifier des particules considérablement hétérogènes dans tous les jeux de données (Extended Data Figs. 3, 4). Paramètres CTF et trajectoires par particule dans les deux meilleures classes du NADP+ les données ont été estimées et corrigées (polissage des particules) à l’aide de RELION 3.036. La résolution locale a été estimée à l'aide de Resmap 5.037.
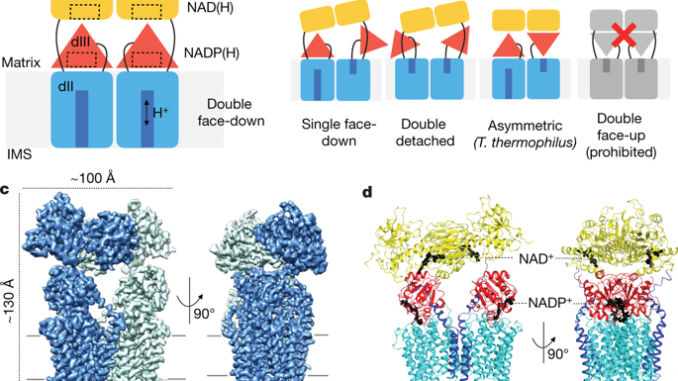
Pour le PNDA+ ensemble de données, nous avons effectué un tour de classification 2D, suivi de deux tours de classification 3D avec des particules triplées et 2x, respectivement, ce qui a donné huit classes contenant 342 284 particules; il en ressort qu'il existe une hétérogénéité considérable dans les conformations de dI2 et dIIIb. Pour remédier à cela, les particules ont été extraites à nouveau sans tri et raffinées avec un dII2dIII2 masque à 3,5 Å puis classé en dix classes sans recherches ni masque pour séparer les particules en fonction de la position relative du dI2. En combinant des classes similaires et en les auto-raffinant, nous avons séparé six états différents de NNT qui diffèrent les uns des autres en ce qui concerne les positions de dI.2 et dIIIb. Deux de ces classes ont été résolues à 13 Å, deux à 7 Å et les deux meilleures classes à 3,7 et 3,2 Å. D'après ces deux dernières cartes, il était évident que la densité de di2 était toujours plus faible que pour le reste de la particule, ce qui indique une hétérogénéité restante dans les positions de dI2; en tant que tels, les cartes finales pour ces deux classes ont été produites en combinant des cartes affinées par focus du dII.2dIII2 et di2 les pièces. Cela a donné la carte finale pour la classe 1 (double face cachée) avec une résolution de 2,9 Å dans le dII2dIII2 et 3.2 Å résolution dans le di2 régions, et pour la classe 2 (simple face cachée), nous avons obtenu une résolution de 3,5 Å pour le dII2Résolution dIII et 4.1 Å pour le dI2 les régions. En raison de la mauvaise qualité de la carte pour la classe unique face cachée dans certaines régions de dIIb (CL6, 7, 8), nous avons suggéré qu'il subsiste une certaine hétérogénéité de conformation dans cette classe et nous avons effectué une autre série de classification de ces particules. avec un masque autour de dIIb pour trier les particules avec dIIIb lié ou semi-attaché à dII. Cette procédure a éliminé 52% des particules et nous avons effectué un auto-raffinement avec un dII2masque dIII des 28 498 particules restantes avec dIIIb détaché sans ambiguïté. Cela a donné une carte de 3,7 Å avec une densité de dIIb bien mieux résolue et clairement différente de la densité de dIIa (qui a dIII attaché), et elle a ensuite été utilisée pour la construction de modèles.
Pour le jeu de données NADPH, nous avons effectué un tour de classification 2D suivi d'un tour de classification 3D sur 510 073 particules triées en grappes avec les mêmes paramètres que dans le NADP.+ ensemble de données; cela n'a abouti à aucune classe comportant des caractéristiques structurelles secondaires visibles. Nous avons effectué des classifications approfondies de ces particules en 4 à 20 classes avec les valeurs du facteur de régularisation. T entre 4 et 12 et le filtrage différent des volumes de référence, qui ont tous échoué à donner une seule classe bien définie avec des caractéristiques structurelles secondaires. Néanmoins, nous avons sélectionné 405 141 particules des 4 meilleures classes d’une classification avec T = 8 et k = 6 classes (les mêmes que pour le NADP+ ensemble de données) pour d’autres tentatives de classification. Une autre série de classifications approfondies a été réalisée sur cet empilement de particules, les meilleurs résultats ayant été obtenus à partir d'une T = 8, k = 20 course. Même dans cette exécution, seules six classes environ environ avaient des fonctionnalités bien définies correspondant aux domaines de NNT. Ces six classes ont été extraites et raffinées pour donner des structures à 8,3, 9,2, 13, 16, 16 et 16 Å, et ont révélé un degré élevé de dI2 et dIII mobilité (Extended Data Fig. 4a). Pour remédier à cela, nous avons essayé d'affiner les 405 141 particules en une seule structure de consensus à 13 Å, suivie d'un raffinement ciblé de l'un ou l'autre.2 ou dII2; Cependant, aucune de ces stratégies n'a donné de structure avec une résolution meilleure que 9 Å, probablement parce que l'hétérogénéité de conformation au sein du raffinement du consensus était trop grande. De plus, les masses moléculaires de dI2 et dII2 ne sont que 80 kDa, et dII2 est masquée par une micelle, ce qui complique encore l'alignement d'un tel ensemble de données hétérogène. Enfin, nous avons effectué le raffinement multi-corps dans RELION 3.0 en partant du raffinement consensus de 405 141 particules et en utilisant des masques pour les deux corps dI.2 et dII2. Ceci a donné une reconstruction de 9 Å de dI2 et une reconstruction de dII de 10 Å2, mais plus important encore, l’analyse en composantes principales a montré un degré élevé de dI2 mobilité dans ces particules (Fig. 2b). Ce n’est pas surprenant, car lors de la dissociation du dIII, dI2 ne reste que faiblement attaché au reste de la particule par un lieur flexible dI – dII.
L'ensemble de données apo-NNT a été entièrement traité dans RELION 3.0. Nous avons sélectionné manuellement 643 bonnes images et extrait 3 × particules en grappes. Une seule conformation de NNT a pu être observée à l'aide de divers schémas de classification 3D. Les 283 706 particules correspondantes ont donc été extraites sans regroupement et ont été auto-raffinées à 4,5 Å. De cette structure, il était clair que dI2 présente la même hétérogénéité que dans le NADP+ ensemble de données. Pour améliorer la résolution dans les dII à dIII, nous avons affiné notre travail autour de cette région et avons classé les particules sans recherche en six classes. La meilleure classe, avec 67 908 particules, a été auto-raffinée et polie pour atteindre une résolution de 3,7 Å. Un polissage ou un sous-classement plus poussé de cette classe n’a pas amélioré davantage la résolution. Tentatives de raffiner la mise au point de dI2 n’a pas amélioré sa résolution par rapport à la structure complète polie des particules sélectionnées finales, dans laquelle la résolution locale en di2 était autour de 5 Å. Nous concluons que dI2 sans nucléotide lié présente encore plus de désordre que lorsque NAD+ est lié, en permettant éventuellement l'échange NAD (H) et dI2 reliure et libération.
Maquette et raffinement
Le modèle initial a été construit dans le double PNAD face cachée+ densité de classe en commençant par la structure cristalline bovine dIII avec une séquence ovine substituée et le modèle d'homologie dI généré à l'aide du serveur Phyre238. Le modèle DII et les lieurs ont été construits manuellement dans Coot39. Les modèles ajustés manuellement ont été raffinés à l'aide de PHENIX Logiciel40 en utilisant une méthode adaptée pour affiner les structures de cryo-EM, qui utilise deux cycles d'un raffinage ADP de groupe, suivis de trois cycles de minimisation globale. Cela permet d'optimiser les facteurs B de sorte que les chaînes latérales de carboxylates endommagées par le rayonnement électronique acquièrent des facteurs B élevés et n'entraînent pas de distorsions de la chaîne principale.41. Le modèle final de NNT est de très haute qualité en termes de géométrie et d’ajustement à la densité (tableau supplémentaire 1).
Des densités pour plusieurs molécules lipidiques ont été observées dans chacune des cavités semi-fermées formées par les hélices TM1, TM2 et TM6 (Extended Data Figs. 5g, 6g, h). Les phosphatidylcholines ont modelé leur structure en se basant sur l’apparence de leur densité et sur le fait que seule la phosphatidylcholine pouvait réactiver un insecte appauvri en lipides NNT42.
Une densité correspondant à la partie adénylée de NAD+ a été observée dans le site de liaison NAD (H) du dI (Extended Data Fig. 5j, k). Nous proposons que cette densité est NAD+ qui sont restés liés à l'enzyme pendant la purification ou, plus probablement, ont été introduits dans l'échantillon en tant que contaminant du NADP+ Solution. Notre PNDA+ était pur à 98% et à 2% de NAD+ contamination, le NAD+ la concentration serait de 100 µM, ce qui est dans la gamme des Kré pour l'enzyme23,28. Cette dernière proposition est étayée par le fait que, dans notre structure, le R139 conservé a la même conformation étendue que dans le Escherichia coli dI2 avec NAD lié+, et diffère considérablement des formes liées à l'apo ou au NADH13. Néanmoins, notre structure est une conformation de NNT à l'état fondamental oxydée de bonne foi, et des travaux biochimiques antérieurs suggèrent que l'échange de nucléotides dans la sous-unité dI peut avoir lieu à n'importe quel stade du cycle catalytique et n'influence pas la conformation globale de l'enzyme.15,16,43,44. Modèles pour le PNDA unique face cachée+ La classe et l'apo NNT étaient basés sur le double NNT face cachée en tant que modèle de départ. Pour apo NNT, en raison du manque d'informations fiables sur la chaîne latérale des acides aminés dans la densité cryo-EM du domaine dI, dI a été modélisé comme un modèle polyalanine basé sur le dI de la structure PDB 6QTI, et a été ajusté à la densité. comme un corps rigide.
Analyse de la structure et préparation des figures
Les scores de conservation évolutifs ont été projetés sur la structure de la transhydrogénase à l'aide de ConSurf45 (Données étendues Fig. 1b). Les molécules d'eau enterrée ont été modélisées à l'aide du logiciel Dowser original.46 (Fig. 3, Données étendues Fig. 7). Le pore de translocation de protons a été prédit et analysé à l'aide d'un serveur MOLEonline47. Les contacts interdomaines et l'énergie de liaison des nucléotides ont été analysés à l'aide du serveur PISA48,49. Les modèles ont été évalués avec MolProbity et EMRinger50,51. Clustal Omega a été utilisé pour l'alignement de séquence52,53. Des modèles et des cartes de densité ont été visualisés à des fins d’analyse et de préparation des figures dans PyMOL 2.2.3 et UCSF Chimera.54.
Mesures d'activité et trypsinolyse
L’activité de transhydrogénation inverse a été mesurée par spectrophotométrie à 30 ° C en suivant la réduction de l’APAD+ (3-acétylpyridine adénine dinucléotide; NAD+ analogue; coefficient d'extinction ε = 5,1 mM−1 cm−1) à 375 nm en utilisant un spectrophotomètre Shimadzu UV-2600 UV-VIS. L'absorption de fond à 455 nm a été soustraite de l'absorption à 375 nm. Le tampon de réaction était similaire au tampon minimal contenant de l'EDTA utilisé dans une publication précédente, avec une modification du détergent et de l'agent tampon pour maximiser l'activité (Tris-HCl 20 mM, pH 6,8, NaCl 50 mM, EDTA 0,5 mM, 0,1% de CHAPS). 0,25 mg ml−1 lipides, NADPH 100 μM et APAD 100 μM+)25. Pour les lipides, nous avons utilisé l’asolectine de soja ou un mélange 4: 1 de 1,2-dioléoyl-sn-glycéro-3-phosphocholine: cardiolipine (DOPC: CL) avec des résultats similaires. La réaction a été démarrée avec l'addition de NNT. L'activité dépend du type de détergent utilisé lors de l'étape finale de chromatographie par exclusion de taille. Les préparations dans le FOM ont donné une activité légèrement supérieure (environ 20 mg−1) que ceux de LMNG (environ 15 U mg−1). Ces valeurs se comparent favorablement aux mesures précédentes25,55.
La trypsinolyse a été réalisée comme décrit précédemment avec quelques modifications15. NNT à 0,05 mg ml−1 dans du tampon GF avec HEPES 1 mM, pH 7,4 a été incubé avec des nucléotides 0,4 mM, de la trypsine (à différents rapports de masse) et un tampon BAT 60 mM (Bis-Tris, acétate, tricine 1: 1: 1) à pH 5-8. Comme le pH optimal de la trypsine est d'environ 8, des rapports de masse compris entre 1: 400 et 1:40 et des temps d'incubation compris entre 30 et 60 min ont été utilisés à différentes valeurs de pH pour obtenir des taux de protéolyse comparables.
Reconstitution de protéoliposomes
Le NNT a été reconstitué en protéoliposomes en utilisant une procédure de dilution au détergent telle que décrite précédemment.25. En bref, du NNT purifié dans du FOM ou du LMNG a été mélangé avec un excès de 500 fois de DOPC solubilisé dans 1% de CHAPS. Après une incubation de 10 minutes sur de la glace, la solution a été diluée 100 fois et incubée dans de la glace pendant 3 à 5 heures avant de mesurer les activités. La réaction a été démarrée par addition de substrat (NADPH et APAD+ à 100 µM). De la 3-chlorophénylhydrazone carbonyle (CCCP; 10 µM) a été ajoutée après 1 min pour découpler le gradient de proton entre protéoliposomes afin d'évaluer la dépendance de la réaction de transhydrogénation à la force motrice des protons. Au bout d'une minute supplémentaire, du CHAPS (0,05%) a été ajouté pour solubiliser les protéoliposomes et donner une activité de transhydrogénation inverse totalement découplée des transhydrogénases dirigées vers l'intérieur et vers l'extérieur.
Résumé du rapport
De plus amples informations sur la conception de la recherche sont disponibles dans le Résumé de la recherche Nature Research lié à cet article.
[ad_2]