[ad_1]
La médecine de précision peut être définie comme «une approche émergente du traitement et de la prévention des maladies qui prend en compte la variabilité individuelle des gènes, de l'environnement et du mode de vie de chaque personne» (voir). Des études du projet de microbiome humain intégrateur (iHMP) des États-Unis fournissent une source de données microbiennes et humaines permettant de suivre l'évolution de deux maladies et de la grossesse, ce qui facilitera la compréhension des microorganismes associés à l'hôte et leurs interactions avec l'hôte humain. . Cette ressource sera d’une grande utilité pour les approches futures de la médecine de précision. Ce travail comprend trois études phares: deux rapports de recherche en La nature, et et par Fettweis et al.. Il y a aussi par le consortium de réseau de recherche iHMPet un autre document de recherche Médecine de la nature par Serrano et al..
La recherche sur les maladies chroniques s'est exclusivement concentrée pendant de nombreuses années sur les cellules et les processus humains. Puis, il y a près de dix ans, les analyses de microbes qui vivent sur et dans le corps (le microbiote) et de leurs génomes collectifs (le microbiome) ont été présentées dans des rapports très médiatisés de groupes tels que MetaHIT (Metagenomics of the European Human Intestinal Tract) et le projet américain sur le microbiome humain (HMP) ont attiré l’attention sur la manière dont le microbiote pourrait également être lié à la maladie humaine. Ces travaux témoignent d'un nouvel élan dans la recherche en microbiologie, motivée par les progrès des outils d'analyse moléculaire et génomique, et qui s'est poursuivie sans relâche depuis.
À la suite de la publication de la première série de documents importants sur le microbiome–, la connaissance des habitants microbiens de notre corps a considérablement augmenté. Au fil du temps, l’échelle des données présentées a augmenté en termes de taille de l’échantillon, de profondeur de la couverture génomique et de complexité du plan expérimental. Des méthodes d'analyse ont été développées et sont adaptées aux défis spécifiques de la manipulation des échantillons de microbiome, y compris le stockage, le transport et l'extraction d'ADN., et ils ont également été conçus pour traiter les complexités statistiques des données de microbiome. Ces avancées ont donné un aperçu des interactions complexes qui se produisent entre nos microbes et notre corps, ainsi que de la façon dont celles-ci changent en matière de santé et de maladie, avec des associations entre les changements microbiens et les maladies signalées pour un large éventail de troubles chroniques courants.
Néanmoins, il reste un grand fossé entre notre compréhension actuelle des modèles microbiens et notre capacité à appliquer ces connaissances pour développer des traitements. Une transition est en cours qui devrait nous permettre de passer de la simple connaissance des associations à la maladie à la compréhension des principaux mécanismes d’action responsables.
Des connaissances insuffisantes entravent cette transition de trois manières. Premièrement, la plupart des études ont porté sur l’identification de la composition spécifique du microbiote humain et, dans une moindre mesure, sur le «potentiel fonctionnel» codé par le microbiome – par exemple, les enzymes codées et les molécules qu’ils pourraient fabriquer. Cependant, l'activité réelle du microbiote, telle que les molécules spécifiques produites ou dégradées à un moment donné, est rarement déterminée par les études actuelles.
Deuxièmement, la plupart de ces études sont transversales, c’est-à-dire qu’elles n’analysent qu’un échantillon par personne. Seuls quelques-uns considèrent l'évolution du microbiote dans le temps et, même dans ce cas, de telles études ont tendance à ne concerner qu'un petit nombre d'individus. Des études qui suivent les changements dans le temps sont nécessaires pour un grand nombre de personnes et pour une multitude de maladies.
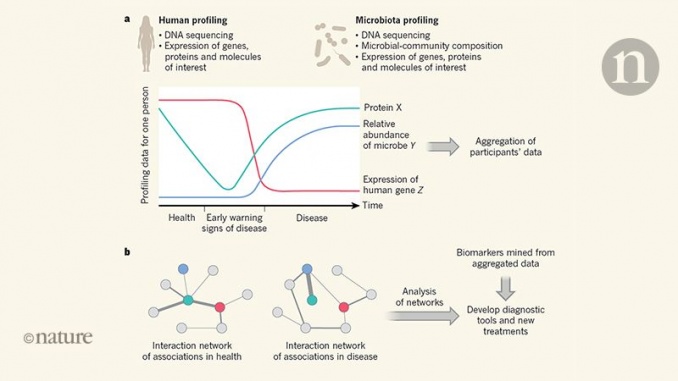
Troisièmement, ces études portent souvent uniquement sur le microbiote humain. Le profilage commun de l'hôte humain et du microbiote est rare, mais négliger cet aspect revient à omettre un élément important du puzzle qui concerne la manière dont nous interagissons avec nos habitants microbiens. Bien que certaines études surmontent une ou deux de ces limitations, les études qui traitent des trois sont rares. C’est là que les données iHMP font la différence.
Deux ans après le travail initial du HMP a été publié en 2012, les National Institutes of Health ont mis en place l'iHMP afin de poursuivre la deuxième phase de cette recherche. Passant de la cartographie du microbiote en santé à la focalisation sur la maladie (Fig. 1), tout en comblant les lacunes susmentionnées en matière de connaissances, les chercheurs participant au pSIh ont abordé trois domaines d’investigation: le début du diabète de type 2., maladie inflammatoire de l'intestin et la recherche de marqueurs de naissance prématurée,.
Ensemble, ces projets ont suivi 463 personnes sur des périodes allant de quelques mois à 4 ans. Les auteurs ont décrit des échantillons fécaux, nasaux ou vaginaux à plusieurs niveaux – capturant les espèces microbiennes présentes et évaluant (en fonction de l’étude) le potentiel fonctionnel du microbiome profilé et les schémas d’expression de ses gènes, protéines et métabolites.
Parallèlement, un profilage des hôtes humains a également été réalisé, comprenant le séquençage des régions codant les protéines de leurs génomes, l'analyse de l'expression génique, la caractérisation de la présence de molécules de signalisation du système immunitaire appelées cytokines et la mesure des niveaux de facteurs cliniquement pertinents tels que le cholestérol. . Bien que le spectre complet des mesures n'ait pas été capturé pour tous les participants à tous les temps, ces études fournissent indéniablement le suivi dynamique le plus complet de l'état de santé de l'hôte humain et des caractéristiques de leur microbiote rapportées jusqu'à présent. La génération de cette riche ressource est une réalisation marquante.
De nombreuses tendances intéressantes ont été observées en termes de variations temporelles du microbiote présentes sur divers sites corporels. L'analyse a révélé que les changements dans la composition et la fonction du microbiote vaginal se produisent principalement pendant le premier trimestre de la grossesse et conduisent à une prédominance de bactéries associées à la santé appartenant au genre. Lactobacilles, en particulier chez les femmes d'ascendance africaine ou hispanique. Fait intéressant, les femmes qui avaient des niveaux inférieurs à la normale de certaines espèces de Lactobacilles étaient plus susceptibles d'accoucher prématurément des bébés. Les personnes atteintes de maladie intestinale inflammatoire présentaient avec le temps un plus haut degré de variabilité dans leur microbiote intestinal que les personnes non atteintes de cette maladie., bien que les molécules dérivées de microbes, telles que les protéines et les métabolites, aient des taux extrêmement variables chez tous les individus étudiés.
Bien que les causes de ces exemples de variation du microbiote dans le temps ne soient pas encore connues, certaines preuves suggèrent que les infections virales sont un déclencheur possible de ce phénomène. Dans les maladies inflammatoires de l'intestin, une augmentation du nombre de virus a parfois été observée juste avant l'apparition d'anomalies dans le microbiote intestinal., qui dans certains cas étaient liés à des épisodes de rechute de la maladie. Dans l'étude de l'apparition du diabète, un échantillonnage intense a été entrepris lorsque les participants présentaient une infection virale du système respiratoire.. Cela a révélé un schéma de changements coordonnés dans le système immunitaire de l'hôte et dans le microbiote intestinal et nasal de participants non diabétiques. Cependant, les participants à risque de développer un diabète avaient une réponse immunitaire altérée et des profils de changement uniques dans leur microbiote. Des études ultérieures seront nécessaires pour évaluer spécifiquement les effets de l'infection virale sur la variation du microbiote au fil du temps et pour déterminer si l'infection virale augmente le risque d'émergence ou de rechute de certaines maladies.
Les papiers iHMP présentent une vaste gamme de corrélations auparavant inconnues entre les composants du microbiote et entre le microbiote et l'hôte. De plus, ce vaste réseau d'associations est cartographié dans le temps. Certaines des connexions les plus intéressantes qui ont été notées dans ces études sont celles qui existent entre des microbes particuliers et la présence de cytokines pro ou anti-inflammatoires. Celles-ci fournissent des indications pour mettre en évidence les relations complexes entre notre système immunitaire et notre microbiote et son activité. Ces associations devront être suivies pour déterminer si des relations directes de cause à effet peuvent être établies.
Même les études portant sur des sujets tels que celles conduites par l'iHMP présentent des faiblesses. Au cours des dernières années, des facteurs clés ayant une incidence sur la composition du microbiote ont été identifiés., et bien que certains, dont l’âge, aient été pris en compte dans les analyses, plusieurs autres facteurs potentiellement cruciaux, tels que le régime alimentaire ou la prise de médicaments, ne l’ont pas été. Des travaux sont nécessaires pour vérifier si les associations identifiées dans les études iHMP sont affectées par ces facteurs. Une autre faiblesse est qu’en raison de l’échelle de temps de l’échantillonnage et de limitations pratiques, la variabilité du microbiote a généralement été étudiée par échantillonnage périodique, avec de longs intervalles entre les échantillonnages. Cependant, pour pouvoir utiliser ces informations en clinique afin de développer des traitements ou des outils de diagnostic, il sera nécessaire de connaître la variabilité au jour le jour.
Un autre problème est que les données générées sont fournies en unités relatives; les abondances bactériennes sont exprimées sous forme de proportion de la population microbienne totale plutôt que sous forme de nombre de cellules. Pour relier avec précision l'activité du microbiote aux processus hôtes, un cadre d'analyse quantitative sera nécessaire pour déterminer expérimentalement les abondances absolues des types de microbes présents dans les échantillons de microbiote.. Enfin, seul un travail minime a été réalisé pour reproduire les résultats du PIDH dans des populations testées indépendamment. Il est de plus en plus évident qu'un large échantillonnage de régions géographiques peut aider à garantir que ces résultats sont robustes et plus largement applicables.
En dépit de ces limitations, l’un des atouts les plus précieux du travail de l’iHMP est sans aucun doute le formidable trésor de données qu’il fournit, dont la plupart sont disponibles gratuitement et qui constituent une ressource utile pour les recherches futures. Ces documents offrent également d’excellents exemples du profilage complet et détaillé d’échantillons prélevés sur des êtres humains, pour lesquels il existe peu de précédents.,. En tant que tels, ces travaux fournissent certaines des données indispensables qui pourraient être utilisées pour former des algorithmes ou pour concevoir des expériences futures. Pour développer des outils cliniques et des traitements dans ce domaine, il sera essentiel de compiler les indications de changement de santé à partir d'une large gamme d'indices capturés à l'aide d'approches à haut débit fournissant des données provenant à la fois des humains et de leurs microbes résidents. Après tout, nous ne sommes pas que des bactéries. Droite?
[ad_2]