[ad_1]
C’est en avril 2019 à l’Allen Institute for Brain Science de Seattle, dans l’État de Washington. Dans une pièce contenant cinq microscopes électroniques à transmission, trois ballons de fête brillants volent. Les ballons célèbrent les chercheurs de l'institut qui ont franchi la dernière étape pour tenter de cartographier chacun des 100 000 neurones et du milliard de connexions, ou synapses, entre eux dans un millimètre cube de cerveau de souris – un échantillon ayant à peu près la taille d'un grain. de sable.
Les microscopes ont fonctionné de manière continue pendant cinq mois, recueillant plus de 100 millions d'images de 25 000 tranches de cortex visuel de souris, chacune d'une épaisseur de 40 nanomètres seulement. Ensuite, les logiciels développés par les informaticiens de l’institut ont mis environ trois mois pour assembler les images en un seul volume 3D. Les bulles indiquent la taille de l'ensemble de données complet, en précisant «2 Po» (2 pétaoctets, ce qui équivaut à 2 millions de gigaoctets) en lettres bleues et argentées. Clay Reid, neurobiologiste à l'Allen, déclare Clay Reid, neurobiologiste à l'Allen, sur plus de trente années d'images satellite de la Terre, recueillies par les missions Landsat, pour environ 1,3 pétaoctet. Institute, citant le poète anglais William Blake.
Le projet millimètre cube cerveau-souris n’est qu’une des nombreuses tentatives menées par diverses espèces pour cartographier un connectome à l’échelle nanométrique – un schéma de câblage du système nerveux comportant des détails au niveau de la synapse. Les neuroscientifiques pensent que ces efforts leur fourniront des informations sans précédent sur la manière dont les circuits neuronaux codent l'information et orientent le comportement – en bref, le fonctionnement du cerveau.
La réalisation ultime dans ce domaine – un connectome à l’échelle nanométrique de tout un cerveau humain – est encore loin. Le cerveau humain a 1015 connexions et contient à peu près le même nombre de neurones qu’il ya des étoiles dans la Voie Lactée, environ 100 milliards. En utilisant les technologies d’imagerie actuelles, il faudrait des dizaines de microscopes travaillant sans relâche, des milliers d’années, pour recueillir les données nécessaires à une telle entreprise.
Mais les progrès de la microscopie, ainsi que le développement d'ordinateurs et d'algorithmes plus puissants pour l'analyse d'images, ont propulsé le domaine de la connectomique à un rythme qui a surpris même les personnes concernées. «Il y a cinq ans, il était trop ambitieux de penser à un millimètre cube», dit Reid. De nombreux chercheurs pensent maintenant que la cartographie de l'intégralité du cerveau de la souris – environ 500 millimètres cubes en volume – pourrait être possible au cours de la prochaine décennie. Et le faire pour le cerveau humain beaucoup plus grand devient un objectif légitime à long terme. «Aujourd'hui, cartographier le cerveau humain au niveau synaptique peut sembler inconcevable. Mais si des progrès constants se poursuivent, tant au niveau des capacités de calcul que des techniques scientifiques, un autre facteur de 1 000 n’est pas exclu. "
Toutes les petites choses
Des connectomes à l'échelle nanométrique ont été complétés chez deux espèces: le nématode Caenorhabditis elegans, en 1986, et la larve d’un organisme marin appelé Ciona intestinalis, en 2016.
Ces cartes neuronales sont un puissant outil de vannage. "Il y a beaucoup d'hypothèses qui ont été réfutées par la C. elegans schéma de câblage », déclare Bobby Kasthuri, neuroscientifique au Argonne National Laboratory de Lemont, dans l’Illinois. Si une observation du système nerveux ou du comportement du ver peut être facilement expliquée par le schéma de câblage, des expériences supplémentaires ne sont pas nécessaires. les chercheurs peuvent passer à des pistes de recherche plus fructueuses. Mais lorsque le connectome n’offre pas une explication facile des résultats, il peut indiquer aux scientifiques des pistes de recherche productives.
Certains chercheurs s'interrogent sur l'intérêt porté aux connectomes à l'échelle nanométrique. Anthony Movshon, neuroscientifique à la New York University à New York, étudie le système visuel, at-il coûté énormément de temps, d'efforts et d'argent à de tels projets. Lorsqu'il est question de cerveaux complexes tels que ceux de souris ou d'humains, «je n'ai pas besoin de connaître les détails du câblage de chaque cellule et de chaque synapse dans chacun de ces cerveaux», explique Movshon. "Ce que j'ai besoin de savoir, ce sont plutôt les principes organisationnels qui les unissent." Ceci, suggère-t-il, peut être assimilé avec un niveau de résolution plus grossier.
Pourtant, le connectome à l'échelle nanométrique est un objectif qui capture l'imagination de nombreux scientifiques. Selon eux, ces travaux pourraient aider à démêler les origines des problèmes de santé mentale et à conduire à des traitements plus éclairés, tout en ayant des applications dans de nombreux domaines, y compris le calcul économe en énergie.
Prolifération de projets
Pour cartographier le connectome nanométrique de C. elegans, dans les années 1980, des chercheurs dirigés par le biologiste Sydney Brenner de l’Université de Cambridge, au Royaume-Uni, ont découpé finement les vers de plusieurs millimètres de long et ont photographié chaque tranche à l’aide d’une caméra de cinéma montée sur un microscope électronique. Dans les images résultantes, ils ont ensuite minutieusement tracé à la main les voies des neurones et leurs connexions.
Mais C. elegans a seulement 302 neurones et environ 7.600 synapses. Les méthodes utilisées pour produire son connectome n’étaient tout simplement pas pratiques à utiliser dans des systèmes nerveux plus importants. Les chercheurs n’ont pas sérieusement envisagé de se lancer dans des projets beaucoup plus vastes qu’en 2004, lorsque le physicien Winfried Denk et le neuroanatomiste Heinz Horstmann, tous deux à l’Institut Max Planck de recherche médicale à Heidelberg, en Allemagne, ont alors proposé l’utilisation d’un microscope automatique pour trancher et imager le cerveau. logiciel pour empiler et aligner les images résultantes.
L'un des plus grands connectomes complets à l'échelle nanométrique à être libérés représente un cube de rétine de souris d'environ 100 micromètres, contenant environ 1 000 neurones et 250 000 synapses. Denk, actuellement directeur de l'Institut de neurobiologie Max Planck à Martinsried (Allemagne), et son collaborateur Moritz Helmstaedter, codirecteur de l'Institut Max Planck de recherche sur le cerveau à Francfort (Allemagne), ont publié l'analyse en 2013. Mais le projet millimètre cube cerveau-souris portera sur 100 000 neurones et d’autres programmes similaires sont également en cours.
«Un millimètre cube est une taille qui semble suffisante, du moins pour les neurones situés au centre de ce grain de sable, pour obtenir la plupart de leurs connexions locales», déclare Nuno da Costa, neuroscientifique à l'Institut Allen. Le projet souris-cerveau permettra donc aux scientifiques d'explorer des circuits locaux complets plutôt que des neurones uniques avec un réseau de connexions clairsemées. Les travaux menés à l'Institut Allen s'inscrivent dans le cadre d'une collaboration avec des chercheurs du Baylor College of Medicine de Houston (Texas), de l'Université Princeton (New Jersey) et de l'Université Harvard de Cambridge (Massachusetts), connus sous le nom de Machine Intelligence from Cortical Networks, financé par le gouvernement américain.
Ses progrès ont conduit certains à prévoir que le connectome à l'échelle nanométrique d'un cerveau de souris complet – susceptible de produire environ un exaoctet (un milliard de giga-octets) de données – pourrait être cartographié au cours de la prochaine décennie. «Cela nécessiterait de nombreux laboratoires», déclare Jeff Lichtman, neuroscientifique à l'Université de Harvard. "Mais c’est faisable", dit-il, "et c’est excitant."
D'autres restent prudents. «Il y a tellement de défis logistiques» pour un projet de cette taille, déclare Stephen Plaza, informaticien au campus Janelia Research du Howard Hughes Medical Institute à Ashburn, en Virginie. Il pense que le champ devrait cibler des projets de taille intermédiaire avant de s'attaquer à quelque chose d'aussi complexe que le cerveau de souris. «Nous sommes encore au stade de la formation à la connectomique», dit-il.
Plaza gère l'un de ces projets. Appelé FlyEM, il vise à produire un connectome du système nerveux central de la mouche du fruit. Drosophila melanogaster. Son équipe s’attend à publier des données sur environ un tiers des D. melanogaster cerveau au début de 2020. Plaza s'attend à ce que le connectome de l'ensemble du système nerveux central – composé d'environ 100 000 neurones et 100 millions de connexions dans le seul cerveau de la mouche, plus un nombre similaire de neurones et de synapses dans le cordon nerveux ventral (à peu près équivalent au la moelle épinière des vertébrés) – suivront quelques années plus tard.
Pendant ce temps, Lichtman travaille sur le poisson zèbre (Danio rerio) connectome, ainsi que l’analyse d’un petit morceau du cerveau humain – un échantillon du gyrus temporal médial, prélevé sur une personne qui subissait une opération du cerveau pour épilepsie. Ce morceau a également un volume d'environ un millimètre cube, mais pour saisir toute l'épaisseur du cortex humain, l'échantillon a la forme d'une dalle plutôt que d'un cube.
Denk et ses collègues cartographient des parties du connectome dans le zébré finch (Taeniopygia guttata), un petit oiseau dont le processus d’apprentissage de la chanson peut donner un aperçu du langage humain. Et Kasthuri a un certain nombre de projets en cours. «Maintenant que de nombreuses données sont disponibles sur les circuits du cerveau de souris, je pense que la meilleure façon de le faire est de regarder les espèces ou le développement», dit-il. "La meilleure information viendra en comparant ce diagramme à d'autres choses."
À cette fin, Kasthuri a pour objectif de cartographier la partie visuelle du cerveau chez des primates non humains, ainsi que chez une pieuvre (Pieuvre bimaculoides). «C’est probablement la créature la plus étrangère qui nous soit encore intelligente, dit-il de la pieuvre. "Donc, je suis intéressé à comparer le câblage de ce cerveau à celui du cerveau de souris."
Kasthuri travaille également sur l'intégralité des connectomes de jeunes souris et de poulpes; comparer ces connectomes immatures à ceux d'animaux adultes pourrait permettre de mieux comprendre comment le cerveau apprend par expérience. En raison de sa petite taille, il espère cartographier le connectome jeune-poulpe dans environ un an.
Espion IA
Maintenant que les chercheurs de l'Institut Allen ont fini d'imager leur millimètre cube de cerveau de souris, ils ont transmis les données à Sebastian Seung, neuroscientifique et informaticien à l'Université de Princeton. Le laboratoire de Seung alignera les images obtenues, puis annotera les synapses et tracera, ou segmentera, les quatre kilomètres estimés de fibres nerveuses contenues dans le volume.
La segmentation a longtemps été l'étape limitante en connectomique. Cela peut prendre des semaines pour tracer à la main le chemin d’un seul neurone dans une pile de micrographies électroniques. Mais maintenant, l'intelligence artificielle s'en mêle. L’équipe de Seung a développé un algorithme d’apprentissage automatique permettant d’évaluer les images pixel par pixel afin de déterminer l’emplacement des neurones.
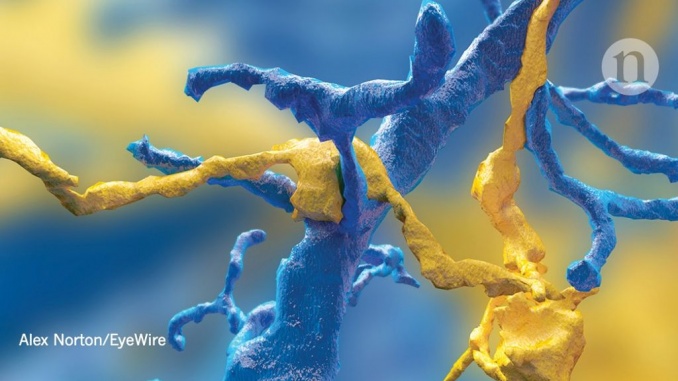
Les ordinateurs peuvent effectuer la segmentation plus rapidement que l’œil humain, ce qui réduit le temps nécessaire au traçage des neurones en quelques minutes ou quelques heures. Mais ils ne sont pas aussi précis: les algorithmes peuvent passer à côté de fragments de neurones ou ne pas fusionner deux neurones en un seul. Les gens sont donc toujours nécessaires pour vérifier la reconstruction. Seung s'attaque à cette exigence par le biais du crowdsourcing et, en particulier, d'un jeu en ligne appelé Eyewire, dans lequel les joueurs sont invités à corriger les erreurs lors du brouillon du connectome. Créée en 2012, Eyewire compte 290 000 utilisateurs enregistrés qui ont collectivement déployé un effort équivalent à 32 personnes travaillant à temps plein pendant 7 ans, explique Amy Robinson Sterling, directrice exécutive de Eyewire.
Jusqu'à présent, les joueurs ont tracé des cellules dans la rétine de la souris. Ils ont contribué à la découverte de six types de neurones, que les joueurs ont choisi de nommer d'après les divinités grecques antiques. Sterling et son équipe préparent une nouvelle version du jeu, appelée Neo, qui sera utilisée avec le jeu de données visual-cortex de souris.
L’interface de Neo est basée sur Neuroglancer, un programme développé par Google qui permet de visualiser des micrographies à plat en noir et blanc, sous forme de forêt 3D colorée de neurones. De nombreux efforts de cartographie de connectome à l'échelle nanométrique utilisent le programme pour visualiser des données.
Google a également développé un algorithme pour la segmentation des neurones. Une équipe dirigée par Viren Jain chez Google AI à Mountain View, en Californie, a conçu un algorithme d’apprentissage automatique appelé réseau de remplissage, qui construit des structures à partir d’un point de l’image, plutôt que de tenter de définir les limites de tous les neurones. immediatement. «C’est un peu comme un humain colorierait un livre de coloriage», déclare Jain. Son équipe applique cette technique aux données FlyEM et a construit un brouillon de connexion d'un cerveau entier de mouches imagé par une autre équipe du Janelia Research Campus. Ils travaillent également avec les données des laboratoires de Denk et de Lichtman.
«C’est vraiment magnifique à regarder», explique Lichtman à propos du résultat, notant que l’algorithme permet de détecter les neurones plus rapidement que son équipe ne peut collecter de données d’imagerie. «Nous ne pouvons pas les suivre», ajoute-t-il. "C’est un endroit formidable."
Jain prend une note plus prudente et souligne que, à mesure que les scientifiques entreprennent des projets de plus en plus vastes, les algorithmes de segmentation doivent devenir plus précis pour que le contrôle humain requis reste réalisable.
Regarder de près
Pendant ce temps, les scientifiques perfectionnent les techniques de microscopie pour produire des images plus nettes et plus détaillées à un rythme beaucoup plus rapide, en prévision de la prise en charge des connectomes à l'échelle nanométrique de grands cerveaux de mammifères.
L'approche conventionnelle de la microscopie en connectomique est un type de microscopie électronique appelé microscopie électronique en coupe série. Les chercheurs incorporent du tissu neural dans du plastique et le découpent en tranches d'une fraction de l'épaisseur d'un cheveu humain. Ils montent ensuite les tranches sur une bande spécialisée et transmettent le résultat – qui ressemble remarquablement à un film sur une bobine – au microscope.
L’avantage de cette méthode est que l’échantillon est préservé et peut être reconfiguré si nécessaire. Mais quelle que soit la précision avec laquelle il est effectué, le découpage de l'échantillon entraîne inévitablement des distorsions qui rendent difficile l'alignement des images.
Une approche plus récente, connue sous le nom de microscopie électronique à balayage à faisceau ionique focalisé (FIB-SEM), utilise un faisceau d'ions chargés pour éliminer une fine couche d'un échantillon de tissu. Le microscope capture une image de la surface fraîchement exposée, puis le processus est répété. L’échantillon FlyEM représente le premier volume important à être imagé par cette méthode.
Bien qu’il manque de rapidité, FIB-SEM a pour avantage que la résolution des images produites est la même dans les trois dimensions, au lieu d’être plus grossière le long de l’axe vertical. Cependant, les échantillons ne peuvent être imagés qu'une seule fois, car ils sont vaporisés au cours du processus. De plus, le champ de vision est très petit, ce qui le rend difficile à appliquer à des échantillons plus grands. (Même le cerveau de la mouche des fruits, qui a à peu près la taille d'une graine de pavot, doit être coupé en morceaux plus petits.) Une méthode appelée microscopie électronique à balayage à faisceau d'ions ioniques (GCIB-SEM), développée par Kenneth Hayworth, neuroscientifique Au Janelia Research Campus, les travaux sont similaires, mais le champ de vision est plus large, ce qui facilite son utilisation pour l'imagerie de grands cerveaux.
GCIB-SEM pourrait également être plus compatible avec les microscopes électroniques multifaisceaux, dont les chercheurs espèrent qu’ils accéléreront l’acquisition des images. De nombreux faisceaux d'électrons balayent un échantillon en même temps, ce qui permet au microscope de capturer des centaines de millions de pixels par seconde. Lichtman utilise une machine de 61 faisceaux produite par Carl Zeiss, et Denk en possède un de 91. Et des microscopes électroniques avec des centaines de faisceaux sont sur le point d'arriver, ce qui pourrait éventuellement capturer un gigapixel de données d'imagerie toutes les secondes.
Donne du sens
Mais la vitesse crée ses propres problèmes. Maintenant que les projets de connectome à l'échelle nanométrique produisent rapidement des données, un autre défi se profile: comment donner un sens à tout cela. «Nous avons tellement de données traitées à portée de main», déclare Reid. «Un grand nombre de scientifiques pourraient découvrir régulièrement de nouvelles choses sur cet ensemble de données. Beaucoup plus que nous pourrions éventuellement embaucher. "
Il y a aussi la question de la liaison des données de connectivité à l'échelle nanométrique avec celles produites par d'autres projets de neuroscience à grande échelle tels que le projet Human Connectome. Cet effort a eu recours à l’imagerie par résonance magnétique pour analyser le cerveau d’environ 1 200 personnes afin de définir des étendues millimétriques de fibres nerveuses reliant des régions du cerveau. Le résultat était une carte connue sous le nom de macroconnectome.
«Le problème le plus important en neuroscience est le problème d'échelle», déclare David Edwards, néonatologue au Kings College de Londres. Il fait partie du projet "Developing Human Connectome", qui termine ses efforts pour scanner le cerveau de centaines de fœtus dans l'utérus, ainsi que celui de bébés à terme et prématurés. «Il y a de grandes choses qui se font à l'échelle macroscopique, à grande échelle, à l'échelle de la population», a déclaré Edwards. "Mais il y a très peu de moyens de les relier."
De nouvelles sources de données, à certains égards plus détaillées que le connectome à l’échelle nanométrique, font également leur apparition. Par exemple, le connectome fournit uniquement des informations sur l'emplacement des synapses, pas sur leur composition moléculaire. «Je vois cela comme un fossé qui doit être comblé», déclare Seth Grant, neuroscientifique moléculaire à l'Université d'Edimbourg, au Royaume-Uni. "Si vous ne pouvez pas faire le pont, vous ne trouverez pas votre chemin vers le génome." Et ces informations génomiques, suggère Grant, seront essentielles pour comprendre comment l'évolution et la génétique régissent le fonctionnement du cerveau.
Entrez le synaptome. Dans un article de 2018, Grant et son équipe ont répertorié un milliard de synapses dans tout le cerveau de la souris., ce qui leur a permis de définir 37 sous-types sur la base de la teneur en protéines, de leur taille et de leur forme, et d’identifier les modèles de sous-types caractérisant diverses régions du cerveau. L'équipe a également commencé à faire correspondre les sous-types avec les connexions qu'ils établissent. «En associant le synaptome au connectome», déclare Grant, «ce sera l'une des prochaines frontières».
[ad_2]