[ad_1]
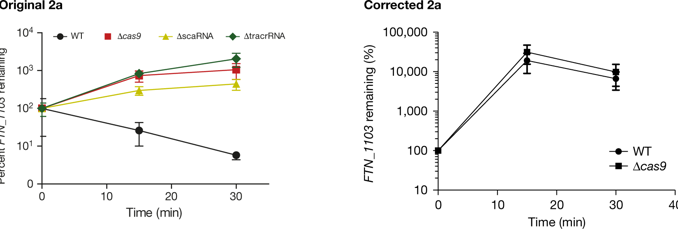
Nous n'avons pas été en mesure de reproduire l'expérience de stabilité de l'ARN de la figure 2a de la présente lettre et nous n'observons plus de diminution, dépendante de Cas9, des niveaux relatifs de l'ARN. FTN_1103 transcription. Les nouvelles données révèlent que le type sauvage et cas9 mutant par délétion (Δcas9) les souches ont des niveaux équivalents de FTN_1103 ARNm à chaque instant (voir Fig. 1 de cet amendement). Nous n’avons pas d’explication sur la divergence avec les données originales, mais cela peut être dû en partie aux complications découlant de la mesure de la stabilité de la FTN_1103 transcrit dans des souches avec des niveaux de base très différents de cet ARNm. Nous n’avons pas non plus été en mesure de reproduire les résultats des expériences d’immunoprécipitation présentées à la Fig. 2g de la Lettre originale et nous n’observons plus de niveaux inférieurs du FTN_1103 transcription après immunoprécipitation avec Cas9 (R59A) –Flag par rapport à Cas9 – Flag de type sauvage (voir Fig. 2 de cet amendement). Nous observons toujours la tendance initiale de niveaux plus élevés de petits ARN (ARNc) associés à CRISPR-Cas et transARN recombinant (tracrRNA) dans l'immunoprécipitation de type sauvage Cas9 – Flag par rapport au mutant Cas9 (R59A) –Flag (Fig. 2e, f de la lettre d'origine). Cependant, nous ne pouvons pas exclure que ces nouveaux résultats reflètent simplement les niveaux globaux de ces transcrits dans les différentes souches, en raison de l’absence biologique d’un contrôle réglementé par Cas9 en dehors de la zone de contrôle. FTN_1103 lieu. Indépendamment de l'expérience d'immunoprécipitation, il existe encore des données génétiques robustes indiquant que l'ARNARN et le tracrARN sont nécessaires pour la répression de Cas9 dépendante de FTN_1103 niveaux d'ARNm.
Stabilité de FTN_1103 ARN dans le type sauvage (WT) et Δcas9 Francisella novicida. Niveaux de FTN_1103 ARN (par rapport à uvrD) ont été mesurés par PCR quantitative (qPCR) 0, 15 et 30 min après le traitement par la rifampicine. Les résultats sont indiqués en pourcentage de FTN_1103 ARNm restant des niveaux à t = 0 (n = 8).
par exemple, qPCR après immunoprécipitation de Cas9 – Flag ou de Casant (R59A) –Flag de type sauvage de lysats cellulaires. Niveaux de ScaRNA (e), tracrRNA (F) et FTN_1103 (g) ont été quantifiés par qPCR par rapport à uvrD (n = 3). Les données sont moyennes et s.e.m. **P ≤ 0,005; ***P ≤ 0,001, étudiant bilatéral t-tester.
De plus, sur la figure 2h de cette lettre, un mutant de tracrRNA (tracrRNA (rc13-17)) contenant des substitutions dans le site d'interaction putatif avec FTN_1103 a été utilisé pour suggérer que l'expression de FTN_1103 a été réprimé via une interaction avec le tracrARN. Bien que non connues à l’époque, les données cristallographiques ultérieures1 indique que la mutation en tracrARN était dans une région importante de la tige-boucle qui interagit avec Cas9, et que Cas9 s'est avéré nécessaire pour la stabilité des taux de tracrARN2. Pour ces raisons, on ne peut plus déduire définitivement le rôle des bases de tracrARN mutées dans la répression de FTN_1103 et n'ont aucune preuve pour soutenir un rôle de tracrARN en interaction directe avec FTN_1103 ARN
Ensemble, ces résultats suggèrent que nous ne disposons pas de preuves pour soutenir la dégradation de l'ARN en tant que mécanisme qui sous-tend la régulation de Cas9 par médiation FTN_1103 expression de l'ARNm. Les autres expériences et conclusions présentées dans la lettre ne sont pas affectées par cet amendement, et nous nous excusons pour les précédentes données trompeuses et pour l’effet qu’elles ont pu avoir sur d’autres personnes sur le terrain. Nous remercions Hannah K. Ratner d’avoir identifié ces erreurs, d’avoir mené les expériences décrites ici avec Siddharth Jaggavarapu et d’avoir rédigé la majorité de cet amendement. La lettre originale n'a pas été corrigée.
1.Deltcheva, E. et al. Maturation de l'ARN CRISPR par le petit ARN trans-codé et le facteur hôte RNase III. La nature 471, 602–607 (2011).
2. Hirano, H. et al. Structure et ingénierie de Francisella novicida Cas9. Cellule 164950–961 (2016).
Informations sur l'auteur
Les affiliations
-
Programme de microbiologie et de génétique moléculaire, Département de microbiologie et d'immunologie, Université Emory, Atlanta, Géorgie, 30329, États-Unis
- Timothy R. Sampson
- Et Anna C. Llewellyn
-
Emory Vaccine Center, Université Emory, Atlanta, Géorgie, 30329, États-Unis
- Timothy R. Sampson
- Anna C. Llewellyn
- & David S. Weiss
-
Centre de recherche sur le primat national Yerkes, Université Emory, Atlanta, Géorgie, 30329, États-Unis
- Timothy R. Sampson
- Anna C. Llewellyn
- & David S. Weiss
-
Division des maladies infectieuses, Département de médecine, École de médecine de l'Université Emory, Atlanta, Géorgie, 30329, États-Unis
- Sunil D. Saroj
- , Yih-Ling Tzeng
- & David S. Weiss
Auteurs
-
Recherche de Timothy R. Sampson en:
-
Recherchez Sunil D. Saroj en:
-
Recherchez Anna C. Llewellyn dans:
-
Recherchez Yih-Ling Tzeng dans:
-
Recherche de David S. Weiss en:
auteur correspondant
Correspondance à
.
Droits et permissions
Pour obtenir la permission de réutiliser le contenu de cet article, visitez RightsLink.
A propos de cet article
Histoire de publication
EST CE QUE JE
commentaires
En soumettant un commentaire, vous acceptez de respecter notre et. Si vous trouvez quelque chose d'abus ou qui n'est pas conforme à nos termes ou directives, veuillez le signaler comme étant inapproprié.
[ad_2]