[ad_1]
Recrutement des participants et consentement de la CISR
Les participants ont donné leur consentement écrit éclairé pour l’étude selon le protocole d’étude de recherche 23602 approuvé par le comité de révision des établissements universitaires de l’Université de Stanford. Tous les participants ont été étudiés après un jeûne nocturne à l’Unité de recherche clinique et translationnelle de Stanford (CTRU). Cette étude est conforme à toutes les réglementations éthiques pertinentes et un consentement éclairé a été obtenu de tous les participants.
Les participants ont été recrutés par le biais d'annonces dans les journaux locaux et les stations de radio recherchant des «volontaires prédiabétiques» à risque de DT2 pour une étude multi-optique longitudinale. Le dépistage dans les CTRU impliquait la collecte des antécédents cliniques, un examen physique, des mesures anthropométriques et des tests sanguins à jeun pour les exclusions, y compris la présence d'anémie définie comme un hématocrite. <30, renal disease defined as creatinine >1.5, antécédents de maladie cardiovasculaire, maligne, inflammatoire ou psychiatrique chronique et antécédents de chirurgie bariatrique ou de liposuccion. Les données de l'étude ont été collectées et gérées à l'aide des outils électroniques de capture de données REDCap hébergés à l'Université de Stanford.
Chaque sujet éligible et ayant donné son consentement a subi une quantification unique de l'absorption de glucose induite par l'insuline via le test de suppression de l'insuline modifié décrit précédemment et validé10,51. En bref, après un jeûne nocturne, les sujets ont été perfusés pendant 180 minutes avec de l’octréotide (0,27 µg / m2 min), insuline (25 mU / m2 min) et du glucose (240 mg / m2 min). On a prélevé du sang à des intervalles de 10 minutes, de 150 à 180 minutes de la perfusion pour mesurer les concentrations de glucose plasmatique (méthode oxymétrique) et d'insuline (dosage radioimmunologique): la moyenne de ces quatre valeurs comprenait les concentrations de SSPG et d'insuline pour chaque individu. À l’état d’équilibre, les concentrations d’insuline (65 μU / ml) étaient similaires chez tous les sujets et le SSPG permettait de mesurer directement la capacité relative de l’insuline à éliminer une charge de glucose: plus la concentration de SSPG était élevée, plus le sujet était résistant à l’insuline. . Tandis que le SSPG est distribué de manière continue, nous avons défini pour cette étude insensible à SPSG <150 mg / dl et insulinorésistant SSPG ≥ 150 mg / dl, en grande partie pour assurer la séparation entre les deux groupes. Dans certains cas, les tests SSPG n’ont pas été effectués, pour des raisons telles que: 1) les participants se sont retirés de l’étude avant les tests prévus; 2) les participants ont choisi de ne pas participer à ce test (c'est-à-dire qu'ils n'y ont pas consenti) en raison de conflits d'horaire ou d'autres raisons personnelles; 3) les participants n'étaient pas éligibles pour le test, comme cela avait déjà été diagnostiqué avec le diabète (la CISR ne le permettra pas).
Préparation des échantillons de sang
Du sang a été prélevé chez des participants ayant jeûné pendant la nuit aux heures indiquées à la CTRU de Stanford. Une aliquote de sang a été incubée à la température ambiante pour se coaguler; Les caillots ont ensuite été sédimentés et le surnageant de sérum a été prélevé à la pipette et immédiatement congelé à -80 ° C. Le sang provenant de tubes d'EDTA séparés a été immédiatement déposé sur du milieu Ficoll et centrifugé par centrifugation sur gradient. La couche supérieure de plasma a été prélevée à la pipette, aliquotée et immédiatement congelée à -80 ° C. La couche de PBMC a été retirée et comptée à l'aide d'un compteur de cellules, et des aliquotes de PBMC ont été ensuite sédimentées et congelées rapidement avec du DMSO / FBS. Pour les analyses multi-omiques subséquentes, les PBMC ont été décongelées sur de la glace, puis lysées et transformées en fractions d’ADN, d’ARN et de protéines en utilisant des colonnes Allprep Spin Columns (Qiagen) conformément aux instructions du fabricant et en utilisant l’option de lyse Qiashredder. L'analyse du plasma a été effectuée sur des aliquotes individuelles afin d'éviter les cycles de congélation / décongélation.
Séquençage de l'exome
En bref, l’ADN a été isolé du sang à l’aide de kits Gentra Puregene (Qiagen) conformément aux instructions du fabricant. Le séquençage de l'exome a été effectué dans un établissement agréé CLIA et CAP à l'aide du test d'exome clinique ACE (Personalis). L’appel de variantes a été effectué à l’aide d’un pipeline automatisé développé en interne.52.
Séquençage d'ARN
Le transcriptome a été évalué en utilisant l'ARN-seq de PBMC congelées en masse. Nous avons utilisé le kit de préparation Qiagen All pour extraire l’ARN total des PBMC conformément au protocole du fabricant. Les banques d’ARN ont été construites à l’aide du kit de préparation d’échantillons TruSeq Stranded RNA LT / HT total (Illumina) contenant 500 ng d’ARN total, conformément aux instructions du fabricant. En bref, l'ARN ribosomal a été appauvri de l'ARN total en utilisant des billes magnétiques Ribo-Zero, puis l'ARN appauvri en ARN ribosomal a été purifié et fragmenté. Une amorce aléatoire avec un adaptateur Illumina a été utilisée pour effectuer une transcription inverse afin d'obtenir une banque d'ADNc. Une séquence d'adaptateur a été ajoutée à l'autre extrémité de la bibliothèque d'ADNc avec une étape de marquage de terminal. La banque d'ADNc a été amplifiée en utilisant les amorces Illumina fournies avec ce kit. La manipulation des liquides a été réalisée avec une plate-forme de manipulation de liquides automatisée Agilent Bravo. Les banques d’ARN-seq ont été séquencées à l’aide de l’instrument Illumina HiSeq 2000 (Illumina) conformément aux instructions du fabricant. Chaque bibliothèque a été quantifiée à l'aide d'un bioanalyseur Agilent et d'une quantification fluorométrique Qubit (ThermoFisher) à l'aide d'un kit haute sensibilité d'ADNdb. Les banques quantifiées, codées par code à barres, ont été normalisées et mélangées à des concentrations équimolaires dans une bibliothèque de séquençage multiplexée. Le séquençage des bibliothèques a été effectué jusqu'à 2 × 101 cycles. Nous avons séquencé en moyenne 5 à 6 bibliothèques par ligne de HiSeq2000. L'analyse des images et les appels de base ont été effectués avec le pipeline Illumina standard.
Le forfait TopHat53 a été utilisé pour aligner les lectures sur le génome de référence hg19 et l'exome personnel, suivies de HTseq et de DESEQ2 pour l'assemblage du transcrit et la quantification de l'expression de l'ARN54,55. Des scripts personnalisés en R et Python ont été utilisés pour les analyses en aval. Pour le prétraitement des données, nous avons d'abord éliminé les gènes avec un nombre de lectures moyen sur tous les échantillons inférieurs à 0,5. Ensuite, les échantillons avec un nombre de lectures moyen sur tous les gènes filtrés inférieurs à 0,5 ont été filtrés.
Le fichier d'entrée contenait 883 échantillons sous forme de colonnes et 25 364 gènes sous forme de lignes. Après les étapes de filtrage, nous avions 883 échantillons avec des données d’expression provenant de 13 379 gènes. Pour les analyses de variance globale et de corrélation, afin de réduire le nombre de caractéristiques, nous avons retiré les gènes dont le nombre moyen de lectures était inférieur à 1, ce qui a donné 10 343 gènes. Nous avons également utilisé l'algorithme xCell56 pour déconvoluer les types de cellules PBMC en fonction des données d'ARN-seq. Pour le calcul des scores d'abondance avec xCell, toutes les données d'expression génétique ont été concaténées dans un seul fichier. Les scores d’abondance ont ensuite été calculés à partir des données d’expression en utilisant xCell (fonction xCellAnalysis exécutée avec l’option ‘rnaseq = TRUE 'et N = 64). Les scores d'abondance des types de cellules PBMC déconvoluées ont ensuite été utilisés pour classifier les événements de stress.
Échantillonnage de microbiome
L'échantillonnage des microbiomes des selles, du nez, de la langue et de la peau a été réalisé conformément au projet de microbiome humain – Protocole d'échantillonnage du microbiome central A (). Une fois les échantillons reçus au laboratoire, ils ont ensuite été stockés à –80 ° C jusqu'à leur traitement ultérieur.
Microbiomique
Extraction d'ADN
L'extraction de l'ADN a été effectuée conformément au protocole A du protocole de prélèvement d'échantillons de microbiome humain du projet de microbiome humain (protocole HMP n ° 07-001, v12.0). L'ADN métagénomique a été isolé dans une hotte propre à l'aide du kit d'extraction d'ADN MOBIO PowerSoil, avec addition de protéinase K, suivi d'un traitement au lysozyme et à la staphylolysine.
Amplification et séquençage de gènes d'ARNr ciblés
Pour l'amplification du gène ARNr 16S (bactérien), les régions hyper-variables V1 – V3 de 16S ont été amplifiées à partir de l'ADN métagénomique en utilisant les amorces 27F et 534R (27F: 5′-AGAGTTTGATCCTGGCTCAG-3 'et 534R: 5'-ATTAGGCGGGGG-3'). . Les oligonucléotides contenant les séquences d’amorce 16S contenaient également une séquence d’adaptateur pour la plate-forme de séquençage Illumina. Une séquence de code à barres unique pour chaque échantillon a été intégrée dans chacun des oligonucléotides direct et inverse utilisés pour créer les amplicons (étiquettes doubles). Les amplicons à code-barres uniques de plusieurs échantillons ont été regroupés et séquencés sur la plate-forme de séquençage Illumina MiSeq en utilisant un protocole de séquençage V3 2 × 300. Le gène de l'ARNr 16S fait environ 1,5 kb et comprend neuf régions variables qui fournissent une grande partie de la distinction de séquence entre différents taxons. Les régions variables 1–3 sont généralement suffisantes pour identifier les taxons jusqu'au niveau du genre et parfois même au niveau de l'espèce. Le logiciel d’Illumina gère le traitement initial de toutes les données brutes de séquençage. Une disparité dans l'amorce et zéro dans les codes à barres ont été appliquées pour affecter des paires de lecture à l'échantillon approprié dans un groupe d'échantillons. Les codes à barres et les amorces ont été supprimés des lectures. Les lectures ont ensuite été traitées en supprimant les séquences de qualité médiocre (qualité moyenne <35) et ambiguës (Ns). Les amplicons chimériques ont été retirés à l'aide d'UChime et les séquences d'amplicons ont été regroupées et les unités taxonomiques opérationnelles (UTO) sélectionnées par la base de données Usearch contre GreenGenes (version de mai 2013). L'affectation taxonomique finale a été réalisée à l'aide du classificateur RDP. Tous les détails ont été exécutés avec QIIME57 avec des scripts personnalisés.
Séquençage métagénomique
L’ADN a également fait l’objet du séquençage métagénomique par canon métagénomique du génome entier pour une sélection de frottis nasaux au cours de l’infection afin d’identifier les bactéries et les virus au niveau de l’espèce (décrit plus en détail dans un document joint39). Les bibliothèques ont été préparées selon un protocole standard d'Illumina et au moins 1 Go de lecture de paires de paires de 150 paires de bases par échantillon a été séquencé sur l'instrument Illumina HiSeq ou MiSeq. Le traitement en aval des lectures mWGS comprenait: a) l’identification et le masquage des lectures humaines (à l’aide de BMTagger du NCBI, ftp://ftp.ncbi.nlm.nih.gov/pub/agarwala/bmtagger); b) élimination des lectures dupliquées qui sont des artefacts du processus de séquençage; c) tailler des bases de mauvaise qualité; et d) le criblage de faible complexité (b – d ont été effectués via PRINSEQ). Les lectures coupées à moins de 60 pb ont été retirées et les lectures de haute qualité restantes ont été analysées en aval.
La métabolomique non ciblée par LC – MS
Les échantillons de plasma ont été préparés et analysés dans un ordre randomisé comme décrit précédemment58. En résumé, les métabolites ont été extraits avec de l'acétone: acétonitrile: méthanol 1: 1: 1, évaporés à sec sous azote et reconstitués dans du méthanol: eau 1: 1 avant analyse. Les extraits métaboliques ont été analysés quatre fois en utilisant une séparation par interaction hydrophile (HILIC) et par chromatographie en phase liquide en phase inverse (RPLC) dans les modes d'ionisation positive et négative. Les données ont été acquises sur un spectromètre de masse Thermo Q Exactive plus pour HILIC et un spectromètre de masse Thermo Q Exactive pour RPLC. Les deux instruments étaient équipés d'une sonde HESI-II et fonctionnaient en mode de balayage complet MS. Les données MS / MS ont été acquises sur des échantillons de contrôle de la qualité (CQ) constitués d'un mélange équimolaire de 150 échantillons randomisés de l'étude. Les expériences HILIC ont été effectuées avec une colonne ZIC-HILIC (2,1 × 100 mm, 3,5 µm, 200 Å; Merck Millipore) et des solvants de phase mobile consistant en acétate d'ammonium 10 mM dans un mélange acétonitrile / eau 50/50 (A) et d'acétate d'ammonium 10 mM. dans 95/5 acétonitrile / eau (B). Les expériences RPLC ont été effectuées en utilisant une colonne Zorbax SBaq (2,1 × 50 mm, 1,7 µm, 100 Å; Agilent Technologies) et des solvants de phase mobile consistant en 0,06% d'acide acétique dans de l'eau (A) et 0,06% d'acide acétique dans du méthanol (B).
Traitement de données métabolomiques
Des extraits métaboliques de 979 échantillons ont été préparés dans un ordre randomisé et les données ont été acquises en trois lots. Les données de LC-MS ont été traitées à l'aide de Progenesis QI (Nonlinear Dynamics). Les caractéristiques métaboliques des blancs et ne présentant pas une linéarité suffisante à la dilution ont été éliminées. Seules les caractéristiques métaboliques présentes dans> 33% des échantillons ont été conservées pour analyse ultérieure et les valeurs manquantes ont été imputées à l'aide de l'outil kméthode des voisins les plus proches. La dérive du signal MS avec le temps pour les données non ciblées ne peut pas être facilement corrigée en utilisant un petit nombre d'étalons internes, car la dérive est non linéaire et dépend du métabolite. Pour contourner ce problème, nous avons appliqué la normalisation LOESS (lissage des diagrammes de dispersion estimé localement) à nos données. Chaque dérive du signal de caractéristique métabolique avec le temps a été corrigée indépendamment en ajustant une courbe LOESS au signal MS mesuré dans les CQ. Les QC ont été injectés tous les 10 échantillons biologiques et consistaient en un mélange équimolaire de 150 échantillons aléatoires de l'étude. Nous avons montré que la normalisation LOESS était efficace pour corriger les dérives de signaux spécifiques aux métabolites intra et inter-lots, comme illustré dans Extended Data Fig. 1e. Après un prétraitement et une annotation supplémentaires des caractéristiques métaboliques, un total de 722 métabolites a été mesuré à l'aide de notre plate-forme de profilage de métabolites, parmi lesquels 431 ont été identifiés en comparant les spectres de temps de rétention et de fragmentation à des standards authentiques ou en comparant les spectres de fragmentation à des référentiels publics.
Protéomique (spectroscopie de masse SWATH)
Les peptides tryptiques des échantillons de plasma ont été séparés sur un système NanoLC 425 (SCIEX). Le débit était de 5 µl / min avec le réglage piège-élution en utilisant ChromXP (SCIEX) 0,5 x 10 mm. Le gradient LC a été réglé sur un gradient de 43 minutes de 4–32% de B pour une durée totale de 1 h. La phase mobile A était composée à 100% d'eau avec 0,1% d'acide formique. La phase mobile B consistait en 100% d'acétonitrile avec 0,1% d'acide formique. Nous avons utilisé une charge de plasma non appauvri de 8 µg sur une colonne ChromXP de 15 cm. L'analyse MS a été réalisée à l'aide de l'acquisition SWATH sur un système TripleTOF 6600 équipé d'une source DuoSpray et d'une électrode ID de 25 µm (SCIEX). Fenêtre variable Q1 Les méthodes d'acquisition SWATH (100 fenêtres) ont été générées en mode MS / MS haute sensibilité avec le logiciel Analyst TF Software 1.7.
Traitement des données protéomiques
Les groupes de pics de séries individuelles ont été statistiquement marqués avec pyProphet59 et toutes les exécutions ont été alignées avec TRIC60 produire une matrice de données finale avec 1% de FDR au niveau du peptide et 10% de FDR au niveau de la protéine. Les abondances en protéines ont été calculées en faisant la somme des trois peptides les plus abondants (méthode top3). Un entretien majeur du spectromètre de masse a entraîné des effets de lot considérables sur les échantillons mesurés. Pour réduire les effets de lot, nous avons effectué une soustraction des composantes principales montrant un biais de lot majeur en utilisant Perseus.61 1.4.2.40.
Essais Luminex
Les taux de cytokines circulantes dans le sang ont été mesurés à l'aide d'un test de capture de billes de conjugaison d'anticorps Luminex à 63 plexes (Affymetrix) qui a été caractérisé de manière approfondie et évalué par le Centre de surveillance immunitaire humain de Stanford (HIMC). Les 63 plex humains ont été achetés à eBiosciences / Affymetrix et utilisés conformément aux recommandations du fabricant, avec les modifications décrites ci-dessous. En bref, les perles ont été ajoutées à une plaque à 96 puits et lavées à l'aide d'une rondelle Biotek ELx405. Des échantillons ont été ajoutés à la plaque contenant les billes mélangées liées à l'anticorps et incubés à température ambiante pendant 1 h, puis incubés pendant une nuit à 4 ° C sous agitation. Les étapes d’incubation à froid et à la température ambiante ont été effectuées sur un agitateur orbital à 500-600 tr / min. Après une nuit d'incubation, les plaques ont été lavées à l'aide d'un laveur Biotek ELx405, puis un anticorps de détection biotinylé a été ajouté pendant 75 min à température ambiante sous agitation. La plaque a été lavée comme ci-dessus et de la streptavidine-PE a été ajoutée. Après incubation pendant 30 minutes à la température ambiante, un lavage a été effectué comme ci-dessus et du tampon de lecture a été ajouté aux puits. Les plaques ont été lues en utilisant un instrument Luminex 200 avec une limite inférieure de 50 billes par échantillon par cytokine. Des billes de contrôle d'analyse personnalisées de Radix Biosolutions ont été ajoutées à tous les puits. Les résultats de différents lots ont ensuite été corrigés à l'aide de contrôles et de réplicats partagés entre les lots. Le dosage a été effectué par Stanford HIMC.
Analyse de données multivariée
Des matrices de données provenant de tous les domaines (test de laboratoire clinique, cytokines, transcriptome, protéome à base de MS, métabolome, données du microbiome 16S et WGS) ont été obtenues et traitées dans un format commun. Les métabolites et les intensités protéiques MS ont été transformés de manière logarithmique,2(n + 1) transformé. Seuls les taxons microbiens qui étaient présents (> 0) ou les gènes microbiens avec une abondance supérieure à 0,1% dans plus de la moitié de la collection (> 400) ont été utilisés plus avant pour l'analyse en aval. Les taxons et gènes microbiens étaient transformés en arcsine pour des analyses linéaires en aval62,63. Les analyses permettant de générer les résultats rapportés sont spécifiées ci-dessous.
Intervalle interquartile de cohorte (IQR) et IQR individuel
Pour évaluer le modèle de dispersion des valeurs par analyte dans l'ensemble de la cohorte et chez chaque sujet, nous avons utilisé l'IQR pour décrire cette variation. Les analytes ont d'abord été normalisés avec un écart-type de 1 centré sur 0 avant d'appliquer IQR (Exp, na.rm = TRUE, type = 8) à partir du package R stats, où Exp correspondait aux valeurs normalisées et transformées de manière linéaire de chaque analyte. Les valeurs de toutes les visites saines de la cohorte entière indépendamment des sujets ont été utilisées pour calculer le RIQ de la cohorte, alors que seules les visites saines du sujet correspondant ont été utilisées pour le RIQ de cette personne. Les courbes de densité de la distribution IQR de chaque ome ont été visualisées par geom_density () dans le paquetage Rggplot2.
Décomposition de la variance de base saine
Comme chaque sujet était échantillonné à plusieurs reprises lors de visites saines, nous avons utilisé des modèles LME pour prendre en compte cette dépendance chez les sujets. Nous avons modélisé des interceptions aléatoires, mais une pente fixe, permettant différents niveaux personnels entre les sujets. Nous avons d’abord transformé linéairement chaque analyte (le cas échéant) et normalisé la variation totale à 1 avant d’appliquer la fonction lmer () du package 'lme4' R, avec la formule suivante: lmer (Exp ~ 1 + jours + A1C + SSPG + FPG + (1 | SubjectID), data = ensemble de données, REML = FALSE), dans laquelle Exp correspond aux valeurs transformées linéairement et normalisées de chaque analyte. Nous avons donc utilisé la corrélation intra-classe (ICC) en tant que proportion de la variation totale expliquée par la structure du sujet dans la cohorte de Vsujet au hasard /Vtotal, dans lequel V était la variance de la composante correspondante extraite par VarCorr () et VLe total était de 1. Les variations expliquées par des facteurs fixes (Days, A1C, SSPG et FPG) ont été extraites par anova ().
Profils MDS sains influencés par un facteur personnel
Les molécules les plus importantes avec la contribution la plus individuelle (la plus élevée en ICC) ou les molécules de chaque molécule ont été utilisées pour une analyse à plus grande échelle multidimensionnelle (MDS). La distance entre les visites saines a été calculée en utilisant la méthode Manhattan dans la fonction metaMDSdist () du package vegan R et l’analyse MDS en utilisant metaMDS () avec k = 2. Nous avons calculé un score d'individualité (ind_score) en utilisant la valeur médiane de lignes de base saines chez chaque individu et en calculant la distance par paire moyenne entre tous les individus parmi les molécules en question. Par conséquent, un ind_score plus élevé et donc une distance moyenne plus grande indique un modèle interpersonnel plus distinct.
Associations avec le temps
Nous avons d’abord utilisé la première visite saine comme base de référence pour chaque sujet. Les changements delta dans les valeurs d'expression lors de visites saines ultérieures ont été utilisés pour établir une corrélation avec le changement delta en jours. Les visites saines de sujets ayant au moins trois visites saines ont été utilisées pour évaluer les associations de temps. Comme chaque sujet était échantillonné à plusieurs reprises lors de visites saines, nous avons utilisé des modèles LME pour prendre en compte cette dépendance chez les sujets. Nous avons utilisé rmcorr64, une méthode proche d’un modèle multiniveau nul à interception variable et d’une pente commune pour chaque individu, et teste en particulier une association commune entre les variables de chaque sujet. Rmcorr calcule une taille d'effet pour représenter de manière appropriée le degré auquel les données de chaque sujet sont reflétées par la pente commune des lignes parallèles les mieux ajustées. La méthode rmcorr adopte une approche méta-analytique et calcule le taux d'erreur (degrés de liberté), P valeur (déterminée par le F-rapport: F(Measure df (1), Error df)) et un intervalle de confiance à 95% des tailles d’effet (IC à 95%). Lorsque la relation entre les variables varie considérablement d’un sujet à l’autre, la taille de l’effet rmcorr sera proche de zéro, avec des intervalles de confiance également proches de zéro. Lorsqu'il n'y a pas de forte hétérogénéité entre les sujets et que les lignes parallèles offrent un bon ajustement, la taille de l'effet rmcorr sera grande, avec des intervalles de confiance serrés. De plus, pour éviter les biais potentiels introduits par les sujets qui avaient trop peu de profils de base, des analyses ont également été effectuées en utilisant un sous-ensemble de 27 sujets ayant plus de 900 jours profilés à comparer. La corrélation des taux a ensuite été corrigée pour les tests d'hypothèses multiples par FDR.
Associations avec le gène SSPG ou différence entre insulinorésistante et insulino-sensible dans un état initial sain
Nous avons utilisé une approche de corrélation inter-individuelle (voir la section ‘Analyse du réseau de corrélation’ ci-dessous). Les valeurs médianes de toutes les lignes de base en bonne santé par sujet ont été utilisées pour associer leurs valeurs SSPG ou pour comparer les résultats insulinorésistants à ceux sensibles à l'insuline. La corrélation de Pearson a été utilisée après transformation linéaire, normalisation et correction de l’IMC, de l’âge et du sexe à l’aide du logiciel R ppcor. Pour la corrélation insulinorésistante / insulino-sensible, la classification insulinorésistante / insulino-sensible a d'abord été appliquée sous forme de variables nominales (insulino-sensible à 0, insulinorésistant à 1) avant d'autres analyses. De plus, avec la méthode de correction de l'IMC, de l'âge et du sexe, nous avons testé la corrélation avec des facteurs de confusion potentiels, tels que le HDL, les triglycérides et le ratio triglycérides / HDL, avec le SSPG.10,15 ainsi que l'état des médicaments sur la statine et tout médicament de contrôle du glucose.
ʻOmics signatures différentielles lors d'événements de stress
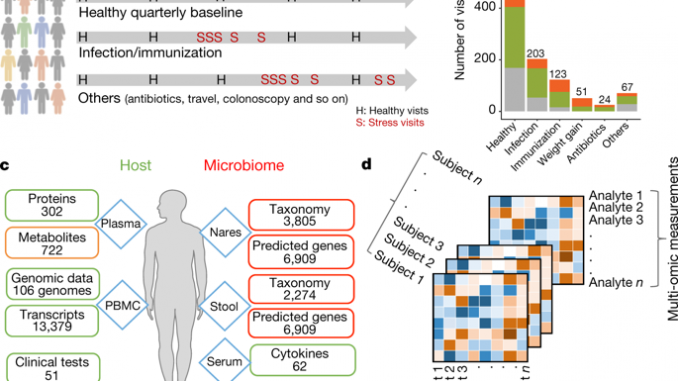
Afin d’identifier les changements temporels dans les molécules ’omics qui s’écartaient de la ligne de base personnelle au cours des événements de stress, nous avons mis en œuvre le test d’aire sous la courbe (AUC). Nous avons défini les catégories longitudinales comme suit: état pré-sain (–H) (valeurs de référence saines dans les 186 jours précédant le début de l'événement), état précoce de l'événement (EE) (visites des jours 1 à 6 de l'événement), événement tardif (EL) état (visites des jours 7 à 14 depuis le début de l'événement), état de récupération (RE) (visites entre les jours 15 à 40 depuis le début de l'événement) et enfin état post-sain (+ H) (visites dans les 186 jours suivant l'événement) ; Fig. 3a).
Le test AUC calcule la somme des moyennes de chaque groupe (EE, EL, RE) après la correction de ligne de base personnelle, la variation de la correction étant également prise en compte. Comme son nom l'indique, l'ASC correspond à l'aire sous la courbe lors de la réponse au stress sur les cinq points temporels classés (–H, EE, EL, RE, + H). Cette quantité peut être interprétée comme la quantité totale de changement d'expression ou d'abondance de molécules s'écartant de la ligne de base personnelle tout au long de la RVI ou de la vaccination.
Pour chaque caractéristique omique, l'hypothèse nulle est que le niveau d'expression moyen reste le même au cours de la RVI ou de la vaccination. Sous l'hypothèse nulle, pour chaque événement de stress, on suppose:
$$ {X} _ {i, alpha} sim N left ({ mu} _ {i}, { sigma} ^ {2} right) $$
pour individuel je et catégorie α∈{EE, EL, RE}. En d'autres termes, le niveau d'expression moyen dépend de l'individu mais pas du type d'événement. Pour chaque échantillon avec une catégorie d'événement Xi, α, α{EE, EL, RE}, la ligne de base personnelle est corrigée en soustrayant la moyenne des points de temps en bonne santé à côté (dans la fenêtre des 186 jours). Laissez l'échantillon corrigé être ({ widetilde {X}} _ {i, alpha} ) et ({ widetilde {{ rm {mean}}}} _ { alpha} soit la moyenne des échantillons corrigés dans le groupe α. Nous utilisons les statistiques de test suivantes:
$$ { rm {AUC}} = frac {{ sum} _ { alpha} { widetilde {{ rm {mean}}}} _ { alpha}} { widetilde {{ rm {std }}}} $$
où ( widetilde {{ rm {std}}} ) est calculée en gardant une trace des poids pour chaque échantillon, de sorte que l’ASC ~N(0, 1) sous l'hypothèse nulle. Puis le P les valeurs peuvent être calculées en conséquence.
De plus, nous avons comparé les performances de la SSC avec l’une des méthodes standard d’analyse longitudinale, à savoir l’analyse de régression linéaire (LR) avec la covariable temporelle. Nous avons utilisé l'analyse LR standard en utilisant l'implémentation Python (statsmodel.OLS)65. Nous avons utilisé le temps comme véritable covariable de valeur et l’ID individuel comme une covariable catégorique. Les méthodes AUC et LR ont bien fonctionné dans l'identification de molécules exprimées de manière différentielle (Extended Data Fig. 4a). Alors que plus de la moitié des caractéristiques identifiées par la méthode LR en tant que molécules exprimées de manière différentielle ont également été trouvées par la méthode AUC (soit 53% pour l'ARN-seq), la méthode AUC a identifié plus de caractéristiques que la méthode LR (Données étendues, Fig. 4a). ). Nous avons donc utilisé le test AUC pour nos analyses différentielles expression / abondance sur les événements de stress.
Afin d’évaluer les changements différentiels pour chaque catégorie d’événement (c’est-à-dire uniquement EE par rapport au niveau de référence personnel), nous avons utilisé t-tester. Le choix du jumelé tLe test consistait à normaliser l’analyse de tous les types de données omiques. Nous avons effectué le test AUC et apparié t-test sur des données pré-traitées, y compris le transcriptome, le métabolome, le protéome, la cytokine, le microbiome intestinal r16S, le microbiome nasal r16S, les gènes prédits microbiens (gènes KO par KEGG) et les résultats de tests de laboratoire clinique. Les données du transcriptome ont été normalisées en fonction du facteur de taille et converties dans l'espace du journal par journal (Xij + 0,5) pour les analyses en aval. Pour la correction du facteur de taille, la moyenne géométrique de l'expression de chaque gène dans tous les échantillons a d'abord été calculée. Le facteur de taille de chaque échantillon est la médiane d’un gène à l’autre du ratio de l’expression à la moyenne géométrique du gène. Ensuite, les nombres lus pour chaque échantillon ont été normalisés par le facteur de taille. La correction du facteur de taille a été effectuée comme décrit dans DESeq255,66.
Nous avons utilisé q-values pour le contrôle de taux de découverte faux. Nous avons considéré des molécules avec q <0,1 pour être significatif. Il est à noter que pour les comparaisons par étape (par exemple, EE versus base individuelle en bonne santé), nous avons comparé la performance du t-test avec la méthode DESeq2 pour des analyses différentielles de données de transcriptome (Extended Data Fig. 4b, tableau complémentaire 39). Le nombre de transcriptions différentiellement exprimées (q <0,1) identifié par apparié t6 857 et le nombre de transcriptions exprimées de manière différentielle (q <0,1) identifié par la méthode DESeq2 était de 4 062, dont 71% se chevauchaient avec des paires t– résultats de test (données étendues, figure 4b). De plus, les analyses d'enrichissement de voies pour les transcrits exprimés de manière différentielle par les deux méthodes ont montré les mêmes résultats d'enrichissement de voies (Tableau supplémentaire 40). Parce que la différence entre les résultats obtenus à partir de la paire t-test et les méthodes Deseq2 pour les transcriptions étaient mineures, nous avons décidé d'utiliser le t-tester nos analyses afin de standardiser l’analyse sur tous les types de données omiques.
Nous avons appliqué l'analyse de la voie d'ingéniosité (IPA)67 rechercher des voies enrichies dans notre liste de molécules omiques exprimées différentiellement. Pour l'analyse de la voie canonique intégrée, les transcrits significatifs, les protéines, les métabolites et les cytokines ont été combinés et utilisés comme fichier d'entrée le long de leurs séquences respectives. P valeurs et statistiques AUC. La statistique AUC est utilisée par l’API pour générer une activité de voie z-des scores pour prédire l'activation ou l'inhibition de voies enrichies. Pour l’analyse des catégories d’événements (c’est-à-dire uniquement EE par rapport à la référence personnelle), nous avons utilisé t-tester P valeurs des molécules omiques significatives et du log2(comptages de lecture normalisée de base) pour les changements d'expression ou d'abondance comme entrée pour les analyses IPA. L'algorithme d'enrichissement IPA utilise deux scores qui abordent deux aspects indépendants des analyses. Le premier est le score d’enrichissement basé sur le test exact de Fisher P valeur. le P La valeur représente l'importance du chevauchement entre les molécules régulées observées et prédites. Le deuxième score est l'activation Z-score, qui est une mesure de prévision pour l'état d'activation ou d'inhibition des régulateurs dans les voies. Veuillez noter que l'activation Z-une valeur de zéro pour les voies ayant une grande P valeurs signifie que l'algorithme IPA n'a pas pu prédire l'activation ou l'inhibition de la voie et des régulateurs67.
En outre, afin de dégager des tendances statistiquement significatives (test de l’ASC q <0,1) ’réponses omiques aux événements de stress, nous avons utilisé la reconnaissance de formes longitudinales en utilisant c-veux dire68 clustering sur toutes les données. Nous avons d’abord utilisé la méthode du coude pour identifier le nombre optimal de grappes dans notre ensemble de données. Les données du transcriptome, du protéome, du métabolome, des cytokines, des tests de laboratoire clinique et des microbiomes intestinaux 16S et 16S nasaux, ainsi que des gènes KO microbiens, ont été standardisées pour Z-cotes pour chaque analyte et soumis à c– signifie regroupement au cours de la RVI ou de la vaccination. Chaque sous-parcelle dans Extended Data Figs. Les figures 5 et 6 représentent un cluster unique et sont codées en couleur sur la base des scores de corrélation d'appartenance. Les principales voies canoniques intégrées (transcriptions, protéines, métabolites, cytokines) et les tendances pour les autres analytes supérieurs (microbiome et tests de laboratoire clinique) sont présentées au-dessus de chaque graphique.
Classification des événements de stress
Afin de prédire les événements de stress (c'est-à-dire l'IVR par rapport aux points temporels sains), nous avons testé plusieurs modèles, parmi lesquels les modèles LR et SVM ont donné les meilleurs résultats. Deux prévisions ont été exécutées: 1) des bases saines par rapport à l'IVR (figure 4c, données étendues, figure 8) et 2) des bases saines par rapport à la vaccination (données étendues, figure 9).
Préparation des données
Nous avons utilisé huit ensembles de données: transcriptome, cytokines, métabolome, protéome, microbiome intestinal r16S, microbiome nasal r16S, types de cellules PBMC déconvolutées et données de tests de laboratoire clinique. Pour les données de transcriptome, nous avons appliqué VST (transformation de stabilisation de variance) de l'algorithme DeSeq255 et utilisé un sous-ensemble de gènes qui sont liés au système immunitaire basé sur des études antérieures56 (Tableau supplémentaire 27). Pour les cytokines, métabolomes, protéomes et données r16S, nous avons corrigé le facteur de taille. Les fonctionnalités avec plus de 100 valeurs manquantes ont été supprimées. De plus, les points temporels sains avec des valeurs de HS-CRP supérieures à 10 ont été écartés. La fonction HS-CRP n'a pas été utilisée pour la prévision, car nous avons déjà utilisé cette information pour filtrer les échantillons. Enfin, nous avons appliqué Z-transformation à toutes les entités afin qu'elles aient des moyennes de 0 et des variances de 1 (sur les points temporels).
Nous avons exécuté LR et SVM pour nos modèles de prédiction, tels qu’implémentés dans le package python sklearn. Pour les deux méthodes, la régularisation l1 est utilisée pour favoriser la faible densité du coefficient appris. Deux expériences de prédiction ont été réalisées: sain versus RVI et sain versus immunisation. Nous avons utilisé uniquement les points temporels d’infection et de vaccination proches de l’apparition de la RVI (groupes EE et EL). Les performances de prédiction ont été évaluées par les courbes de caractéristiques de fonctionnement du récepteur (ROC) et les aires sous la courbe de ROC (AUC) pour chaque ome (transcriptome, métabolome, protéome, cytokines, types de cellules PBMC, taxa microbiens intestinaux r16S et taxa microbiens nasaux r16S). tests de laboratoire) et de tous les patients combinés (ou multi-omes). Le graphique ROC montre le taux positif réel (TPR) par rapport au taux de faux positifs (FPR). La courbe a été calculée en faisant varier le seuil de décision pour obtenir différents compromis TPR – FPR.
Pour chaque expérience, nous avons sélectionné au hasard 70% des données pour l'ensemble d'apprentissage et 30% des données pour l'ensemble d'essai. Les points temporels d'un même individu ont été utilisés dans un seul des deux ensembles. Cela a été répété 100 fois. The regularization parameters were selected based solely on the training set, as follows. For each regularization parameter C over the set (0.1, 0.5, 1, 2, 3, 5, 10), the training data were further split into train_train and train_test. A classification model (LR or SVM) was trained on train_train and evaluated on train_test. This was done five times and the regularization parameter with the smallest error on train_test was chosen as the optimal parameter. Then, the model was trained with the entire training set and the optimal regularization parameter, and evaluated on the testing set.
Correlation network analysis
Given the Simpson’s paradox in correlational analyses (wherein trends can disappear or reverse when data sets are combined)69, we used two statistical approaches to investigate between-individual and within-individual correlations separately, as these reveal different perspectives in understanding the associations.
Within-individual correlations (at the personal level)
This takes all the healthy visits per subject into account, and used linear mixed effect models to account for repeated samplings from the same subject. We used the rmcorr method64, which is close to a null multilevel model of varying intercept and a common slope for each individual, and specifically tests for a common association between variables within each subject. Healthy visits were first grouped into insulin-sensitive and insulin-resistant, and each analyte was linearly transformed before applying the rmcorr() function from the rmcorr R package as explained above (see ‘Associations with time’). As this method relies on repeated measures within each subject and specifically tests for a common association between variables within each subject, potentially confounding factors between subjects, such as sex, age and BMI, do not apply, which is in contrast to the between-individual correlation method below.
Between-individual correlations (at the cohort level)
This first takes the median value of all healthy visits per subject, linearly transforms and then corrects for sex, age and BMI before applying the regression pcor.test() function from the ppcor R package. As this method replies on the median value of repeated measures within each subject, those become independent observations presenting different subjects, so this is suitable for standard linear regression methods downstream.
P values obtained from the above two approaches were further multiple hypothesis corrected by the total number of pairwise comparisons using the FDR method as implemented by p.adjust(p.value, method = “fdr”) in R. We used q < 0.05 as the significant cutoff for all ’omic analytes.
For microbiome-related networks, we implemented two approaches to construct a correlational network that accounts for the compositionality effect. In the first approach, we used centered log ratio (CLR)70 as a preprocessing transformation method that addresses compositionality in microbial data71. Given a sample with ré taxa, the CLR transformation can be obtained as follows:
$$begin{array}{l}{x}_{{rm{CLR}}}=left({rm{log}}left({x}_{1}/Gleft(xright)right),{rm{log}}left({x}_{2}/Gleft(xright),….,{rm{log}}left({x}_{D}/Gleft(xright)right)right)right),\ Gleft(xright)=sqrt(D){{x}_{1}{x}_{2}{x}_{3}… {x}_{D}}end{array}$$
Because microbial taxa span different taxonomic levels (phylum, class, order, family, genus), we used CLR transformation on each taxonomic level separately. After accounting for compositional effect via CLR transformation, we addressed the intra-personal correlation of repeated measurements in the calculation of correlation coefficient between taxa by using repeated measures correlation (rmcorr) method64. Hence, by using this approach, we accounted for compositional effects via CLR and repeated measurements via rmcorr (CLR + rmcorr). For all microbiome–host networks (Fig. 5c, d), we used this approach to calculate the correlation to host ’omics (transcriptomics, proteomics, metabolomics, cytokines, and clinical data).
In the second approach, we used SparCC72 to construct a microbial–microbial network over repeated measurements73 (Fig. 5a, b). We used the python implementation of SparCC in with parameters: –iter = 20,–xiter = 10,–threshold = 0.1. Also, we obtained the P value for each correlation coefficient by bootstrapping the data set 100 times and applying SparCC to each of those 100 data sets. We applied SparCC to features from each taxonomic rank separately to avoid correlations between parent and child taxa. We further compared the microbiome–microbiome network calculated either by SparCC or by CLR+rmcorr (Supplementary Table 32). Correlation coefficients obtained by either method were linearly associated, and more than 50% of significant correlations overlapped, indicating that both methods generally agree. However, some correlations were detected by only one method. Future studies are needed to improve the microbial correlation techniques, as also suggested previously73.
Outlier analysis
To account for healthy baseline variability, only subjects with at least three healthy visits were included in the analysis. Z-scores were calculated for each analyte after log2-transformation using the median value among healthy visits for each subject. Outliers were defined as being in the 95th percentile of Z-score distribution for each analyte. The outlier proportion across assays was calculated by normalizing the number of outliers from each assay to the total number of analytes profiled with the corresponding assay. The percentage of outlier analytes across assays in each participant was then normalized to 100%. Analytes with more than 50% of missing values or zeros were discarded. For transcriptomics data, we arbitrarily chose to discard genes with low expression (log2 normalized read count <5 in more than 50% of the subjects).
While providing tremendous amounts of molecular information, our study has several limitations that necessitate future investigation. First, it is possible that protein isoforms and/or transcript variants are also critical for accurately evaluating host states, which were not examined in the current study. Our studies of microbial changes are also limited, as microbial profiles are based on 16S sequencing, which only allows taxonomy assignments at the genus level and thereby limits more precise interpretations requiring species or strain classifications. Second, as our study is observational, samplings in our study are both planned (most healthy visits) and spontaneous (some healthy visits and most stress visits), resulting in uneven collections. In addition, we utilized a variety of techniques to profile different molecules, so each type of data set has its inherent errors specific to its corresponding platforms. As such, our data are heterogenous by nature, and require custom and novel methods that statistically account for many sources of variation. Our analyses here tend to account for some sources of variation, but additional methods are necessary for future work. Last, we only considered BMI, age and sex as the universal confounding factors in the current study for our correlational analyses. However, there may be additional factors, such as diet and exercise, that need to be considered. For instance, in our analysis of age associations, we cannot exclude the possible influence of changes in lifestyle over the profiling period. Nonetheless, as our cohort is currently expanding to include more participants with continuing longitudinal samplings and archived biobanked collections, we believe it will provide a rich and valuable resource for future research both experimentally and informatically.
Reporting summary
Further information on research design is available in the Nature Research Reporting Summary linked to this paper.
[ad_2]