[ad_1]
Recrutement et prélèvement d'échantillons
Recrutement
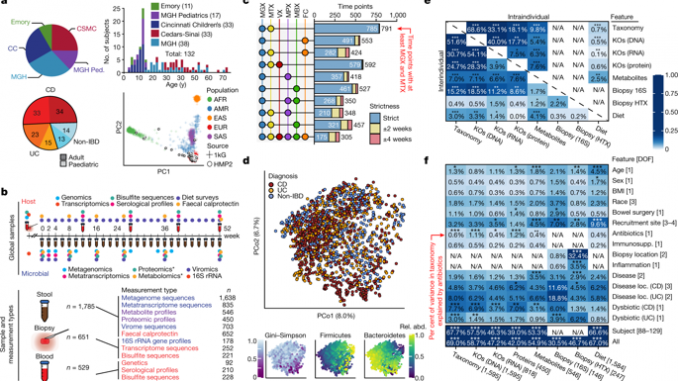
Cinq centres médicaux ont participé à l’IBDMDB: l’hôpital pour enfants de Cincinnati, l’hôpital Emory University, l’hôpital général du Massachusetts, l’hôpital général du Massachusetts pour enfants et le centre médical Cedars-Sinai. Les patients ont été approchés pour être recrutés sur rendez-vous pour le dépistage systématique du cancer colorectal lié à l'âge, le traitement d'autres symptômes gastro-intestinaux (GI), ou une suspicion de MICI, avec une imagerie positive (par exemple, un épaississement de la paroi du côlon ou une inflammation iléale) ou des symptômes de maladies chroniques. diarrhée ou saignements rectaux. Les participants n'auraient pas pu subir un dépistage préalable ou une coloscopie diagnostique. Les participants potentiels ont été exclus s'ils ne pouvaient ou ne voulaient pas fournir de tissus, de sang ou de selles, s'ils étaient enceintes, s'ils présentaient un trouble de la coagulation ou une infection gastro-intestinale aiguë, s'ils étaient activement traités pour une tumeur maligne par chimiothérapie, s'ils avaient reçu un diagnostic indéterminé. une colite ou une chirurgie gastro-intestinale majeure antérieure telle qu'une déviation iléale / colique ou un j-pouch. Lors du recrutement, une coloscopie initiale a été réalisée pour déterminer les strates de l'étude. Les sujets non diagnostiqués porteurs d’une MII sur la base de résultats endoscopiques et histopathologiques ont été classés comme «témoins non-MII», y compris les individus sains susmentionnés se présentant pour un dépistage de routine, et ceux présentant des symptômes plus bénins ou non spécifiques. Cela crée un groupe témoin qui, sans être complètement «en bonne santé», diffère des cohortes de MICI spécifiquement par son statut clinique. Les différences observées entre ces groupes sont donc plus susceptibles de constituer des différences spécifiques aux MII, et non des différences imputables à une détresse générale de l'IG. Au total, 132 sujets ont participé à l'étude (Extended Data Table 1).
Conformité réglementaire
L'étude a été examinée par les commissions d'examen institutionnel de chaque site d'échantillonnage: coordination globale des données des partenaires (IRB # 2013P002215); Cohorte d'adultes en HGM (CISR no 2004P001067); MGH Paediatrics (IRB # 2014P001115); Emory (N ° IRB00071468); Centre médical de l’hôpital pour enfants de Cincinnati (2013-7586); et centre médical Cedars-Sinai (3358 / CR00011696). Tous les participants à l'étude ont donné leur consentement éclairé écrit avant de fournir des échantillons. Chaque CISR a une assurance à l’échelle fédérale et suit les règles établies dans 45 CFR Partie 46. L’étude a été menée conformément aux principes éthiques énoncés dans la Déclaration de Helsinki et aux exigences des réglementations fédérales applicables.
Prélèvement et conservation des échantillons
Des échantillons destinés à la recherche (biopsies, prélèvements sanguins et échantillons de selles) ont été recueillis au cours de la coloscopie de dépistage, à raison de cinq visites de suivi trimestrielles au dispensaire (appelées «situation initiale», visite 2, etc., au cours du mois 0, 3, 6, 9 et 12) et toutes les deux semaines par courrier.
Biopsies
Les biopsies ont été principalement recueillies lors de la première coloscopie de dépistage, où environ quatre à quatorze biopsies ont été collectées pour chaque sujet. Pour chaque site échantillonné (au moins iléon et à 10 cm du rectum, plus des sites d'inflammation facultatifs), une biopsie a été prélevée pour l'histopathologie standard dans l'établissement d'échantillonnage, deux biopsies ont été collectées et stockées dans RNAlater pour la génération de données moléculaires (hôte et microbiologique). conservés à –20 ° C) et une biopsie a été prélevée et placée dans un tube stérile contenant 5% de glycérol (conservé à –80 ° C). Si possible, des biopsies supplémentaires à partir de tissu enflammé et de tissu non enflammé à proximité ont été prélevées chez des participants atteints de MC ou de CU. Pour les adultes, une deuxième série de biopsies a également été collectée à chaque emplacement (rectum et iléon) pour la culture de cellules épithéliales (pour les protocoles détaillés, voir). Toutes les biopsies ont été conservées jusqu'à deux mois sur le site de collecte et expédiées toute la nuit sur de la glace carbonique vers la Washington University pour la culture de cellules épithéliales ou vers le Broad Institute pour le profilage moléculaire.
Échantillons de sang
Des échantillons de sang (sang total et sérum) ont été prélevés lors des visites cliniques trimestrielles. Pour le sang total, 1 ml de sang a été recueilli et stocké à –80 ° C. Pour le sérum, le sang a été prélevé dans un tube SST de 5 ml et laissé à température ambiante pendant 40 min. Celui-ci a été centrifugé pendant 15 minutes à 3 000 tr / min. et des portions de 0,5 ml ont été immédiatement aliquotées dans des microtubes de 2 ml. Les tubes ont été conservés à –80 ° C.
Échantillons de selles
Les échantillons de selles ont été collectés lors des visites cliniques et toutes les deux semaines par courrier en utilisant un kit de collecte à domicile développé pour le projet () et préalablement validé46. Les participants ont d'abord déposé leurs selles dans un bol de récupération suspendu au-dessus d'une commode. Ils ont ensuite collecté deux aliquotes à l'aide d'une pelle pour transférer les selles dans deux tubes Sarstedt 80.623: l'un contenant environ 5 ml d'éthanol à 100% de biologie moléculaire et l'autre sans conservateur. Des échantillons de selles ont ensuite été envoyés par chaque participant par FedEx au Broad Institute, où ils ont été traités immédiatement avant le stockage à –80 ° C. Le tube d'éthanol a été centrifugé pour former un culot sous-aliquoté et le surnageant a été transféré dans un nouveau tube pour analyse métabolomique. Les selles de l’éthanol ont été réparties dans des tubes cryogéniques de 2 ml en aliquotes de 100 à 200 mg, en donnant la priorité aux échantillons pour le séquençage méta’omique, la métabolomique et la viromique dans cet ordre. Les selles restantes ont été stockées dans des tubes aliquotes supplémentaires. Cent milligrammes de selles sans éthanol ont été stockés pour le dosage de la calprotectine fécale et le reste a été conservé dans un second tube. Tous les échantillons ont été stockés à -80 ° C après réception avant traitement. Cette méthode de collecte à domicile produisait auparavant des résultats reproductibles par rapport aux échantillons surgelés46, cohérent avec les observations précédentes sur tous les types de données47,48,49. Notez qu'une estimation précise de la teneur en eau dans les selles n'a pas pu être obtenue car les échantillons ont été collectés par les sujets et conservés dans de l'éthanol à la température ambiante jusqu'à ce que des aliquotes soient générées pour les différentes plateformes de génération de données.
Participant et exemple de métadonnées
Les descriptions de chaque participant et de l'échantillon ont été capturées au départ et accompagnant chaque collection d'échantillons, respectivement. Au départ (c'est-à-dire pendant ou avant la coloscopie de dépistage), les sujets ont rempli un questionnaire sur les symptômes signalés, le questionnaire sur la maladie inflammatoire de l'intestin court.50, un questionnaire sur la fréquence des repas et un questionnaire sur l'environnement, ainsi que le score endoscopique simple51 pour les sujets de CD ou Score de Baron52 pour les sujets UC a été évalué.
Au cours des deux visites de suivi et des échantillons de selles envoyés par la poste, les sujets ont rempli un questionnaire sur l’indice d’activité et un rappel alimentaire pour évaluer leur indice d’activité (HBI pour CD ou SCCAI pour UC) et rappeler rétrospectivement leur régime alimentaire récent. Tous les questionnaires, ainsi que les protocoles détaillés (y compris les numéros de produits), peuvent être consultés sur le portail de données de l’IBDMDB à l’adresse. Les réponses et les métadonnées sont disponibles sur et les résumés des phénotypes pour les échantillons et les sujets sont fournis (Fig. 3 supplémentaire), ainsi que des résumés des séries chronologiques finales pour chaque sujet (Fig. 2 supplémentaire).
Traitement des échantillons de selles
Selection d'Echantillon
La sélection de l'échantillon s'est déroulée en deux phases, avec un premier cycle de génération de données produisant un ensemble pilote de données métagénomiques et métatranscriptomiques, qui a été analysé séparément.53. Cet échantillon pilote comprenait au moins un échantillon par participant inscrit à l’étude à ce moment-là, deux cures de longue durée par groupe de maladies (CD, UC, non-IBD) et plusieurs cures de plus courte durée, soit 300 échantillons. Pour un sous-ensemble de 78 échantillons, des données métatranscriptomiques ont été générées. Les échantillons ont été choisis sur la base de leur masse, en sélectionnant préférentiellement des échantillons pouvant être re-séquencés si nécessaire au cours de la génération ultérieure de données.
Pour la deuxième phase, la plus vaste de la génération de données, des échantillons de selles ont été sélectionnés pour différents tests dans le but de générer des données couvrant autant d’aspects de la cohorte que possible, y compris des cours par sujet, des points de temps globaux pour les sujets et des échantillons à partir de. tous les patients, phénotypes, tranches d'âge, centres cliniques, etc. (Fig. 1b). Le sous-ensemble de mesures effectuées pour chaque échantillon a été déterminé en grande partie par les besoins en aliquotes (en particulier, les besoins en masse pour le dosage par rapport à la quantité fournie par le patient) et le coût.
Pour la protéomique et la métabolomique, six points temporels globaux ont été équitablement répartis dans la série chronologique d'une année sur autant de sujets que possible. Des restrictions telles que la masse d'échantillon disponible et les échantillons manquants ont été incorporées en sélectionnant l'échantillon approprié le plus proche dans le temps, ce qui a entraîné de légères irrégularités dans le modèle d'échantillonnage. Au total, 546 profils de métabolites et 450 profils de protéomique ont été générés. Parmi ces échantillons, 768 ont été sélectionnés pour la métagénomique, la métatranscriptomique et la viromique, ce qui correspond à 8 plaques de 96 échantillons chacune. Les échantillons déjà sélectionnés pour la protéomique ou la métabolomique ont été hiérarchisés pour faciliter l'analyse intégrée des données (316 échantillons avaient une masse suffisante), donnant ainsi six points de temps globaux pour tous les sujets. Dans les cas où l'échantillon respectif n'était pas disponible pour un sujet, l'échantillon approprié le plus proche dans le temps a été sélectionné. Les sujets présentant des fluctuations plus importantes de leurs scores HBI ou SCCAI ont ensuite été hiérarchisés pour un échantillonnage plus dense, donnant lieu à 12 cours de longue durée pour 5 participants atteints de CD, 4 à la CU et à 3 sans IBD. La sélection comprenait également 23 réplicats techniques pour la métagénomique, la métatranscriptomique et la viromique.
Enfin, 576 échantillons supplémentaires ont été sélectionnés spécifiquement pour le séquençage métagénomique (6 plaques), soit un total de 1 344 échantillons métagénomiques. La priorité a été donnée aux échantillons à des points temporels globaux préalablement sélectionnés et à des parcours longs, limités par la masse disponible pour d'autres types de mesure. Ce processus a ajouté quatre points de temps globaux supplémentaires, ainsi que 15 cours de longue durée (représentant 10 participants avec CD, 10 avec UC et 7 sans IBD), et 22 échantillons précédemment séquencés pour les données pilotes et représentés. répétitions techniques supplémentaires. Enfin, 522 échantillons ont été sélectionnés pour les mesures de calprotectine fécale, en donnant la priorité aux échantillons sélectionnés pour toute autre génération de données multi-omiques et en représentant une vue d'ensemble de la cohorte. Sur un total de 2 653 échantillons de selles prélevés, 1 785 ont généré au moins un type de mesure (figure 1b).
La sélection d'échantillons pour le séquençage d'ARN-seq et 16S à partir de biopsies, et le génotypage d'hôte à partir de prélèvements sanguins, visaient à couvrir les 95 sujets ayant fourni au moins 14 échantillons de selles, comme le permettaient la disponibilité de biopsies et de prélèvements sanguins pour chaque test. La sélection d'échantillons à partir de biopsies visait en outre à couvrir les biopsies de sites enflammés et non enflammés. Au total, 254 biopsies ont été sélectionnées pour l'ARN-seq, couvrant 43 participants atteints de MC, 25 atteintes de CU et 22 sans MII, et réparties sur les sites de biopsie et les états inflammatoires (Extended Data Fig. 6A); et 161 biopsies ont été sélectionnées pour le séquençage 16S, couvrant 36 participants avec CD, 21 avec UC et 22 sans IBD. Le séquençage de l'exome a été effectué chez 46 participants atteints de CD, 24 patients atteints de CU et 22 patients sans MICI.
La sélection des échantillons pour les types d'échantillons restants (RRBS, sérologie sanguine) comprenait tous les échantillons avec un échantillon approprié disponible.
Tests de séquençage
Isolement de l'ADN et de l'ARN pour la métagénomique et la métatranscriptomique
L'acide nucléique total a été extrait d'une partie aliquote de chaque échantillon de selles testé via le Chemagic MSM I avec le Chemagic DNA Blood Kit-96 de Perkin Elmer. Ce kit combine une lyse chimique et mécanique avec une purification à base de billes magnétiques. Avant l'extraction sur le MSM-I, du tampon TE, du lysozyme, de la protéinase K et du tampon RLT avec du bêta-mercaptoéthanol ont été ajoutés à chaque échantillon de selles. La solution de lysat de selles a été mélangée au vortex.
Les échantillons ont ensuite été placés sur l'unité MSM I pour automatiser les étapes suivantes: des billes magnétiques M-PVA ont été ajoutées à la solution de lysat de selles et brassées au vortex. L'acide nucléique total lié à la perle a ensuite été retiré de la solution en utilisant une tête magnétique à 96 barres et lavé dans trois tampons de lavage à base d'éthanol. Les billes ont ensuite été lavées dans un tampon final de lavage à l'eau. Enfin, les billes ont été plongées dans du tampon d'élution pour remettre en suspension l'échantillon d'ADN en solution. Les billes ont ensuite été retirées de la solution, en laissant l'éluat d'acide nucléique total purifié. L'éluat a ensuite été divisé en deux volumes égaux: l'un pour l'ADN et l'autre pour l'ARN. La solution SUPERase-IN a été ajoutée aux échantillons d'ADN et la réaction a été nettoyée à l'aide de billes AMPure XP SPRI. De la DNase a été ajoutée aux échantillons d'ARN et la réaction a été nettoyée à l'aide de billes AMPure XP SPRI.
Les échantillons d'ADN ont été quantifiés à l'aide d'un test PicoGreen basé sur la fluorescence. Les échantillons d'ARN ont été quantifiés à l'aide d'un test RiboGreen basé sur la fluorescence. La qualité de l'ARN a été évaluée par analyse de frottis sur le Caliper LabChip GX.
Séquençage du métagénome
Des métagénomes ont été générés à partir de l’ADN résultant pour 1 638 échantillons de selles, sélectionnés pour obtenir à la fois un aperçu général des points temporels alignés ciblés pour tous les sujets (Fig. 1b), complétés par un échantillonnage dense de sujets ayant tendance à avoir une plus grande activité de la maladie, déterminé par leurs scores HBI ou SCCAI.
Les banques de fragments de génome entier ont été préparées comme suit. Les échantillons d’ADN métagénomique ont été quantifiés par le dosage d’ADNdb PicoGreen de Quant-iT (Life Technologies) et normalisés à une concentration de 50 pg / ul. Des banques de séquençage Illumina ont été préparées à partir de 100 à 250 pg d’ADN à l’aide du kit de préparation de bibliothèque d’ADN Nextera XT (Illumina) conformément au protocole recommandé par le fabricant, les volumes de réaction étant mis à l’échelle en conséquence. Avant le séquençage, les bibliothèques ont été regroupées en collectant des volumes égaux (200 nl) de chaque bibliothèque à partir de lots de 96 échantillons. Les tailles et les concentrations d'insertions pour chaque bibliothèque regroupée ont été déterminées à l'aide d'un kit Agilent Bioanalyzer DNA 1000 (Agilent Technologies). Les bibliothèques ont été séquencées sur HiSeq2000 ou 2500 2×101 pour donner environ 10 millions de lectures finales appariées. Le démultiplexage et la génération post-séquençage des fichiers BAM et FASTQ ont été générés à l'aide de la suite Picard ().
Séquençage du métatranscriptome
Des métatranscriptomes ont été générés pour 855 échantillons de selles, sous-échantillonnés à partir de sélections métagénomiques comme ci-dessus. Les banques d'ADNc d'Illumina ont été générées à l'aide d'une version modifiée du protocole RNAtag-seq54. En bref, 500 ng-1 µg d'ARN total ont été fragmentés, débarrassés de l'ADN génomique, déphosphorylés et ligaturés à des adaptateurs d'ADN portant des codes à barres 5′-AN8-3 'de séquence connue avec un groupe bloquant 5' et 3 '. Les ARN codés à barres ont été regroupés et appauvris en ARNr en utilisant le kit d'appauvrissement en ARNr RiboZero (Epicentre). Les pools d'ARN codés par code à barres ont été convertis en banques d'ADNc d'Illumina en deux étapes principales: (i) transcription inverse de l'ARN en utilisant un amorce conçue pour la région constante de l'adaptateur avec code à barres avec addition d'un adaptateur à l'extrémité 3 'de l'ADNc par matrice commutation à l'aide de SMARTScribe (Clontech) comme décrit55; (ii) L'amplification PCR utilisant des amorces dont les extrémités 5 'ciblent les régions constantes des adaptateurs 3' ou 5 'et dont les extrémités 3' contiennent les séquences complètes d'Illumina P5 ou P7. Les banques d’ADNc ont été séquencées sur la plate-forme Illumina HiSeq2500 pour générer environ 13 millions de lectures de bouts appariées.
Viromics
Nous avons sélectionné 703 échantillons de selles pour le profilage viral, en suivant la sélection de l'échantillon utilisée pour la métatranscriptomique et ajustés légèrement uniquement lorsque des aliquotes n'étaient pas disponibles (Fig. 1c). Les acides nucléiques viraux ont été extraits à l'aide du kit d'isolement d'ARN viral MagMax (AM1939, Thermo Fisher Scientific). L'ARN viral a été soumis à une transcription inverse à l'aide de SuperScript II RT (18064014, Thermo Fisher) et d'hexamères aléatoires. Après une courte élimination des molécules et de l'hexamère de manière aléatoire à l'aide de ChargeSwitch (CS12000, Thermo Fisher), les molécules ont été amplifiées et marquées avec une construction BC12-V8A256 utilisant la polymérase AccuPrimeTM Taq et nettoyé avec le kit ChargeSwitch.
Les amplicons viraux résultants ont été normalisés, regroupés et transformés en une bibliothèque Illumina sans cisaillement. La bibliothèque (150 à 600 pb) a été chargée dans un Illumina HiSeq 2000 (Illumina, Carlsbad, CA) et séquencée à l'aide de la chimie 2 × 100 pb. Les lectures ont été démultiplexées dans un échantillon en utilisant les codes-barres préfixant read-1 et read-2, ce qui permet de ne faire aucune différence. Les lectures démultiplexées ont ensuite été traitées en supprimant les codes à barres, les séquences d’amorces semi-aléatoires et les adaptateurs Illumina. Ce processus utilisait un démultiplexeur personnalisé et l'algorithme BBDuk inclus dans BBMap (). L'ensemble de données découpées résultant a été analysé à l'aide d'un pipeline créé au Centre de métagénomique et de recherche sur le microbiome d'Alkek au Baylor College of Medicine.57. En résumé, le pipeline d'analyse virale utilise un algorithme de classification pour créer des génomes viraux putatifs à l'aide d'une stratégie d'assemblage de cartographie qui exploite les informations d'alignement des nucléotides et traduites. Les taxonomies virales ont été attribuées à l'aide d'un système de notation incorporant les résultats de l'alignement des nucléotides et des nucléotides traduits par base et optimisé pour le rang taxonomique à la résolution la plus élevée.
Métabolomique
Sélection d'échantillons, réception et stockage
La sélection des échantillons pour la métabolomique visait à obtenir uniquement un large échantillon de nombreux sujets. Au total, 546 échantillons de selles ont été sélectionnés pour le profilage (Fig. 1b). Une partie de chaque échantillon de selles sélectionné (40 à 100 mg) et le volume total du conservateur d'éthanol d'origine ont été stockés dans des tubes à centrifuger de 15 ml à -80 ° C jusqu'à ce que tous les échantillons aient été collectés.
Traitement des échantillons
Les échantillons ont été décongelés sur de la glace puis centrifugés à 4 ° C, 5000g) pendant 5 min. L'éthanol a été évaporé à l'aide d'un léger courant d'azote gazeux à l'aide d'un évaporateur d'azote (TurboVap LV; Biotage) et stocké à -80 ° C jusqu'à ce que tous les échantillons de l'étude aient été séchés. Des homogénats aqueux ont été générés par sonication de chaque échantillon dans 900 pi de H2O en utilisant un homogénéisateur à sonde à ultrasons (Branson Sonifier 250) réglé sur un cycle de travail de 25% et un contrôle de sortie de 2 pendant 3 min. Les échantillons ont été conservés sur la glace pendant le processus d'homogénéisation. L’homogénat de chaque échantillon a été aliquoté en deux échantillons de 10 µl et deux échantillons de 30 µl dans des tubes à centrifuger de 1,5 ml pour la préparation d’échantillons LC – MS et 30 µl d’homogénat de chaque échantillon ont été transférés dans un tube conique de 50 ml sur créer un échantillon de référence regroupé. Le mélange de référence rassemblé a été mélangé au vortex, puis aliquoté (100 µl par aliquote) dans des tubes à centrifuger de 1,5 ml. Des aliquotes et des échantillons de référence ont été stockés à -80 ° C jusqu'à la réalisation d'analyses LC – MS.
Analyses LC – MS
Une combinaison de quatre méthodes LC-MS a été utilisée pour profiler les métabolites dans les homogénats fécaux, comme précédemment publié58; deux méthodes qui mesurent les métabolites polaires, une méthode qui mesure les métabolites de polarité intermédiaire (acides gras et acides biliaires, par exemple) et une méthode de détermination du profil lipidique. Pour la file d'attente d'analyse dans chaque méthode, les sujets ont été randomisés et les échantillons longitudinaux de chaque sujet ont été randomisés et analysés en tant que groupe. De plus, des paires d'échantillons de référence regroupés ont été insérées dans la file d'attente à des intervalles d'environ 20 échantillons pour le contrôle de la qualité et la normalisation des données. Des échantillons ont été préparés pour chaque méthode à l'aide de procédures d'extraction adaptées aux conditions de chromatographie. Les données ont été acquises à l'aide de systèmes LC – MS composés de systèmes Nexera X2 U-HPLC (Shimadzu Scientific Instruments) couplés à des spectromètres de masse Q Exactive / Exactive Plus (Thermo Fisher Scientific). Les détails de la méthode sont résumés ci-dessous.
Méthode LC – MS 1: HILIC-pos (analyses MS en mode ionique positive de métabolites polaires). Des échantillons de LC-MS ont été préparés à partir d’homogénats de selles (10 µl) par précipitation de protéines avec addition de neuf volumes d’acétonitrile / méthanol / acide formique à 74,9: 24,9: 0,2 v / v / v contenant des étalons internes marqués à l’isotope stable (valine d8). , Isotec et phenylalanine-d8, Cambridge Isotope Laboratories). Les échantillons ont été centrifugés (10 min, 9 000g, 4 ° C), et les surnageants ont été injectés directement dans une colonne Atlantis HILIC de 150 × 2 mm (Waters). La colonne a été éluée de manière isocratique à un débit de 250 µl / min avec 5% de phase mobile A (formiate d’ammonium 10 mM et 0,1% d’acide formique dans l’eau) pendant 1 min, suivie d’un gradient linéaire jusqu’à 40% de phase mobile B (acétonitrile avec 0,1% d’acide formique) en 10 min. Les analyses MS ont été effectuées en utilisant une ionisation électrospray en mode ion positif en utilisant une analyse complète m/z 70–800 à une résolution de 70 000 et un taux d’acquisition de données de 3 Hz. Les autres paramètres MS sont les suivants: tension de pulvérisation ionique, 3,5 kV; température capillaire, 350 ° C; sonde de température de chauffage, 300 ° C; gaz de gaine, 40; gaz auxiliaire, 15; et le niveau RF de la lentille S 40.
Méthode 2 de LC – MS: HILIC-neg (analyse de MS par métabolisme polaire en mode ion négatif). Des échantillons de LC – MS ont été préparés à partir d'homogénats de selles (30 µl) par précipitation de protéines, en ajoutant quatre volumes de méthanol à 80% contenant de l'inosine-15N4, de la thymine-d4 et du glycocholate-d4 (Cambridge Isotope Laboratories). Les échantillons ont été centrifugés (10 min, 9 000g4 ° C) et les surnageants ont été injectés directement sur une colonne Luna NH2 de 150 x 2,0 mm (Phenomenex). La colonne a été éluée à un débit de 400 ul / min dans des conditions initiales de 10% de phase mobile A (acétate d'ammonium 20 mM et d'hydroxyde d'ammonium 20 mM dans de l'eau) et de 90% de phase B mobile (hydroxyde d'ammonium 10 mM en 75:25). v / v acétonitrile / méthanol) suivi d’un gradient linéaire de 10 min en phase mobile à 100% A. Des analyses MS ont été effectuées en utilisant une ionisation par électrospray en mode ion négatif en utilisant une analyse complète m/z 60–750 avec une résolution de 70 000 et un taux d’acquisition de données de 3 Hz. Les autres paramètres MS sont les suivants: tension de pulvérisation ionique, –3,0 kV; température capillaire, 350 ° C; sonde de température de la sonde, 325 ° C; gaz de gaine, 55; gaz auxiliaire, 10; et le niveau RF de la lentille S 40.
Méthode LC – MS 3: C18-neg (analyse en mode ions négatifs de métabolites de polarité intermédiaire, par exemple, acides biliaires et acides gras libres). Les homogénats de selles (30 µl) ont été extraits avec 90 µl de méthanol contenant du PGE2-d4 comme étalon interne (Cayman Chemical Co.) et centrifugés (10 min, 9 000 pi).g4 ° C). Les surnageants (10 µl) ont été injectés sur une colonne ACQUITY BEH C18 de 150 x 2,1 mm (Waters). La colonne a été éluée de manière isocratique à un débit de 450 ul / min avec 20% de phase mobile A (0,01% d'acide formique dans de l'eau) pendant 3 min, suivie d'un gradient linéaire en 100% de phase mobile B (0,01% d'acide acétique dans de l'acétonitrile). plus de 12 min. Les analyses MS ont été effectuées en utilisant une ionisation électrospray en mode ion négatif en utilisant une analyse complète m/z 70–850 avec une résolution de 70 000 et un taux d’acquisition de données de 3 Hz. Les autres paramètres MS sont les suivants: tension de pulvérisation ionique, –3,5 kV; température capillaire, 320 ° C; sonde de température de chauffage, 300 ° C; gaz de gaine, 45; gaz auxiliaire, 10; et niveau RF de la lentille S 60.
LC-MS Méthode 4: C8-pos. Les lipides (polaires et non polaires) ont été extraits des homogénats de selles (10 µl) en utilisant 190 µl d'isopropanol contenant du 1-dodécanoyl-2-tridécanoyl.sn-glycéro-3-phosphocholine comme étalon interne (Avanti Polar Lipids; Alabaster, AL). Après centrifugation (10 min, 9 000g, température ambiante), les surnageants (10 µl) ont été injectés directement dans une colonne ACQUITY BEH C8 de 100 × 2,1 mm (1,7 µm; Waters). La colonne a été éluée à un débit de 450 µl / min de manière isocratique pendant 1 minute à 80% de phase mobile A (95: 5: 0,1 v / v / vl d’acétate d’ammonium 10 mM / méthanol / acide acétique), suivie d’un gradient linéaire. à 80% de phase mobile B (99,9: méthanol / acide acétique à 0,1 v / v) en 2 min, un gradient linéaire à 100% de phase B mobile pendant 7 min, puis 3 min à 100% de phase B mobile. Des analyses MS ont été effectuées. en utilisant ionisation électrospray dans le mode ion positif en utilisant l'analyse complète du balayage sur m/z 200 à 1 100 avec une résolution de 70 000 et un taux d’acquisition de données de 3 Hz. Les autres paramètres MS sont les suivants: tension de pulvérisation ionique, 3,0 kV; température capillaire, 300 ° C; sonde de température de chauffage, 300 ° C; gaz de gaine, 50; gaz auxiliaire, 15; et niveau RF de la lentille S 60.
Traitement de données métabolomiques
Les données brutes LC – MS ont été acquises sur l'ordinateur d'acquisition de données interfacé avec chaque système LC – MS, puis stockées sur un système de stockage de fichiers robuste et redondant (Isilon Systems) accessible via le réseau interne du Broad Institute. Les données non ciblées ont été traitées à l'aide de Progenesis QIsoftware (v 2.0, Nonlinear Dynamics) pour détecter et désisotopes, aligner le temps de rétention chromatographique et intégrer les zones de pic. Les pics d'identifiant inconnu ont été suivis par méthode, m/z et temps de rétention. L'identification des pics de LC-MS de métabolites non ciblés a été réalisée: i) en faisant correspondre les temps de rétention et les masses mesurés aux mélanges de métabolites de référence analysés dans chaque lot; et ii) l'appariement d'une base de données interne de plus de 600 composés caractérisés à l'aide des méthodes du Broad Institute. La dérive temporelle a été surveillée et normalisée en fonction de l’intensité des caractéristiques mesurées dans les échantillons de référence regroupés.
Protéomique
Sélection d'échantillons et LC – MS / MS
La sélection des échantillons pour la protéomique a largement suivi la sélection des échantillons pour la métabolomique (Fig. 1b, c), avec de légers ajustements lorsque des aliquotes n'étaient pas disponibles. Au total, 447 échantillons de selles ont été ciblés pour le profilage. À partir des échantillons sélectionnés, les protéines ont été digérées de manière protéolytique à l'aide de trypsine, et chaque produit de digestion a été soumis à un fractionnement automatisé hors ligne en phase inverse à pH élevé avec concaténation de fractions. L'analyse LC – MS / MS de chaque fraction a été réalisée à l'aide d'un spectromètre de masse Thermo Scientific Q-Exactive Orbitrap de UCLA, équipé d'une interface nano-ESI construite sur mesure. Les échantillons ont été chargés sur une colonne LC interne capillaire garnie (taille de particule de 70 cm x 75 µm, 3 µm) et les données ont été acquises pendant 120 minutes. Des spectres MS précurseurs ont été recueillis sur 400 à 2 000 m/z, suivi des spectres MS / MS dépendant des données des douze ions les plus abondants, en utilisant une énergie de collision de 30%. Un temps d'exclusion dynamique de 30 s a été utilisé pour discriminer les ions analysés précédemment.
Identification des peptides et synthèse des données sur les protéines
Les spectres de masse issus des analyses obtenues ont été évalués à l'aide du MSGF.+ Logiciel59 v10072 en utilisant les génomes de référence intestinaux HMP 1 (HMP_Refgenome-gut_2015-06-18). En bref, après la conversion des assemblages métagénomiques en cadres de lecture ouverts prédits (par exemple, des protéines prédites), des banques ont été créées en utilisant les directions avant et arrière pour permettre la détermination du FDR. La base de données de leurres inversés permet de mesurer le taux de détection de faux résultats, ce qui permet à son tour de calculer le FDR et de filtrer les données de manière appropriée afin de maximiser les identifications de peptides réels tout en minimisant les identifications fausses. MSGF+ a ensuite été utilisé pour rechercher les données expérimentales de spectres de masse dans les bases de données de leurres inversés et inversés. Seuil pour les données incluses: MSGF+ spectres de probabilité (> 1 × 10dix, équivalent à un BLAST e valeur de masse), précision de la masse (± 20 p.m), taux de protéine FDR de 1% et identification d’un peptide unique par protéine.
Calprotectine fécale
La calprotectine fécale a été quantifiée pour 652 échantillons de selles, qui ont été stockés à –80 ° C sans conservateur avant traitement. La sélection de l'échantillon s'est concentrée sur l'obtention d'une vaste enquête sur tous les sujets plutôt que sur des séries chronologiques détaillées (Fig. 1b). La calprotectine a été quantifiée à l’aide du test ELISA QUANTA Lite Calprotectine (Inova Diagnostics 704770), conformément au protocole du fabricant. Entre 80 et 120 mg de selles ont été utilisés pour la saisie. Le temps d'incubation avant l'arrêt de la réaction a été ajusté pour obtenir la DO405 valeurs dans la plage suggérée pour le test.
Traitement des échantillons de biopsie
Co-isolement de l'ADN et de l'ARN à partir de tissu congelé
L'ADN et l'ARN ont été extraits de biopsies conservées ultérieurement avec l'ARN à l'aide du kit AllPrep DNA / RNA Universal de Qiagen. Des échantillons biologiques ont été coupés en morceaux de 20-25 mg sur un lot de neige carbonique, puis placés dans des tubes avec une perle d’acier pour une homogénéisation mécanique et un tampon hautement dénaturant contenant de l’isothiocyanate de guanidine, qui inactive immédiatement les DNases et les RNases pour assurer l’isolation de l’ADN intact. et l'ARN. Après homogénéisation, le lysat a été passé sur une colonne de centrifugation AllPrep DNA Mini. Cette colonne, en combinaison avec le tampon à haute teneur en sel, permet une liaison sélective et efficace de l'ADN génomique. La digestion de la protéinase K sur colonne dans des conditions de tampon optimisées permet la purification de hauts rendements en ADN de tous les types d'échantillons. La colonne a ensuite été lavée et l'ADN a été élue dans du tampon TE. La colonne de centrifugation AllPrep DNA Mini a été digérée par la protéinase K en présence d'éthanol. Cette digestion optimisée, associée à l'addition ultérieure d'éthanol supplémentaire, a permis une liaison appropriée de l'ARN total, y compris le miARN, à la colonne de centrifugation RNeasy Mini. Les échantillons ont ensuite été digérés avec de la DNase I pour garantir des rendements élevés en ARN sans ADN. Les contaminants ont été efficacement éliminés et l'ARN élué dans l'eau.
Profilage du gène de l'ARNr 16S
Nous avons sélectionné 178 biopsies pour le profilage taxonomique basé sur l’amplicon 16S. Le protocole de séquençage des gènes de l'ARNr 16S a été adapté du projet Earth Microbiome60 et le projet de microbiome humain61,62,63. En bref, l'ADN génomique bactérien a été extrait de la masse totale des spécimens biopsiés à l'aide du kit d'isolement de l'ADN MoBIO PowerLyzer pour tissus et cellules et de spatules stériles pour le transfert de tissu. La région 16S VNA de l'ADNr a été amplifiée à partir de l'ADN extrait par PCR et séquencée dans la plate-forme MiSeq (Illumina) en utilisant le protocole à paires appariées de 2 x 250 pb, donnant des lectures d'extrémités de paires qui se chevauchaient presque complètement. Les amorces utilisées contenaient des adaptateurs pour le séquençage MiSeq et des codes-barres à index unique, de sorte que les produits de PCR puissent être regroupés et séquencés directement.61, ciblant au moins 10 000 lectures par échantillon.
Les paires de lecture ont été démultiplexées et fusionnées à l'aide de USEARCH v7.0.109064. Les séquences ont été regroupées en OTU à un seuil de similarité de 97% en utilisant l'algorithme UPARSE65. Les OTU ont ensuite été mappés sur un sous-ensemble de la base de données SILVA66 contenant uniquement des séquences de la région V4 du gène de l’ARNr 16S pour déterminer les taxonomies. Les abondances ont ensuite été récupérées en mappant les lectures démultiplexées sur les UPUSE OTU, produisant les profils taxonomiques finaux. Les 150 échantillons avec au moins 1 000 lectures cartographiées ont été utilisés dans les analyses en aval.
Host RNA-seq
Construction d'une bibliothèque d'ADNc
Au total, 252 biopsies ont été sélectionnées pour le profilage transcriptionnel. L'ARN total a été quantifié à l'aide du kit de dosage d'ARN RiboGreen de Quant-iT et normalisé à 5 ng / µl. Après étalement, 2 µl de contrôles ERCC (avec une dilution au 1/1 000) ont été introduits dans chaque échantillon. Une portion aliquote de 200 ng pour chaque échantillon a été transférée dans une préparation de banque, qui était une variante automatisée du kit de préparation d'échantillons d'ARNm d'Illumina TruSeq Stranded. Cette méthode préserve l'orientation du brin du transcrit d'ARN. Il utilise des billes d'oligo dT pour sélectionner l'ARNm de l'échantillon d'ARN total. Il est suivi d'une fragmentation par la chaleur et d'une synthèse d'ADNc à partir de la matrice d'ARN. L’ADNc résultant de 500 pb passe ensuite par la préparation de la bibliothèque (réparation finale, addition de base «A», ligature d’adaptateur et enrichissement) en utilisant des adaptateurs indexés conçus par Broad Institute, remplacés par le multiplexage. After enrichment the libraries were quantified using Quant-iT PicoGreen (1:200 dilution). After normalizing samples to 5 ng/μl, the set was pooled and quantified using the KAPA Library Quantification Kit for Illumina Sequencing Platforms. The entire process is in 96-well format and all pipetting is done by either Agilent Bravo or Hamilton Starlet.
Illumina sequencing
Pooled libraries were normalized to 2 nM and denatured using 0.1 N NaOH before sequencing. Flowcell cluster amplification and sequencing were performed according to the manufacturer’s protocols using either the HiSeq 2000 or HiSeq 2500. Each run was a 101-bp paired-end with an eight-base index barcode read. Data were organized using the Broad Institute Picard Pipeline which includes de-multiplexing and lane aggregation.
Blood specimen processing
Serological analysis
We analysed 210 serum samples for expression of ANCA, ASCA, anti-OmpC, and anti-CBir1 by ELISA as previously described67,68. Antibody levels were determined and the results expressed as ELISA units (EU/ml), which are relative to laboratory standards consisting of pooled, antigen-reactive sera from of patients with well-characterized disease.
DNA isolation from whole blood
DNA was extracted using Chemagic MSM I with the Chemagic DNA Blood Kit-96 from Perkin Elmer. The kit combines chemical and mechanical lysis with magnetic bead-based purification. Whole blood samples were incubated at 37 °C for 5–10 min to thaw. The blood was then transferred to a deep well plate with protease and placed on the Chemagic MSM I. The following steps were automated on the MSM I.
M-PVA magnetic beads were added to the blood and protease solution. Lysis buffer was added to the solution and vortexed to mix. The bead-bound DNA was then removed from solution using a 96-rod magnetic head and washed in three ethanol-based wash buffers to eliminate cell debris and protein residue. The beads were then washed in a final water wash buffer. Finally, the beads were dipped in elution buffer to resuspend the DNA. The beads were then removed from solution, leaving purified DNA eluate. The resulting DNA samples were quantified using a fluorescence-based PicoGreen assay.
Host exome sequencing
Ninety-two host exomes were sequenced from DNA extracts using previously published methods69. Whole-exome libraries were constructed and sequenced on an Illumina HiSeq 4000 sequencer with 151-bp paired-end reads. Output from Illumina software was processed by the Picard pipeline to yield BAM files containing calibrated, aligned reads.
Library construction
Library construction was performed as described69 with some slight modifications. Initial genomic DNA input into shearing was reduced from 3 µg to 50 ng in 10 µl solution and enzymatically sheared. In addition, for adaptor ligation, dual-indexed Illumina paired end adapters were replaced with palindromic forked adapters with unique 8-base index sequences embedded within the adaptor and added to each end.
In-solution hybrid selection for exome enrichment
In-solution hybrid selection was performed using the Illumina Rapid Capture Exome enrichment kit with 38 Mb target territory (29 Mb baited). The targeted region includes 98.3% of the intervals in the Refseq exome database. Dual-indexed libraries were pooled into groups of up to 96 samples before hybridization. The liquid handling was automated on a Hamilton Starlet. The enriched library pools were quantified using PicoGreen after elution from streptavadin beads and then normalized to a range compatible with sequencing template denature protocols.
Preparation of libraries for cluster amplification and sequencing
Following sample preparation, the libraries prepared using forked, indexed adapters were quantified using quantitative PCR (purchased from KAPA biosystems), normalized to 2 nM using the Hamilton Starlet Liquid Handling system, and pooled by equal volume using the Hamilton Starlet Liquid Handling system. Pools were then denatured using 0.1 N NaOH. Denatured samples were diluted into strip tubes using the Hamilton Starlet Liquid Handling system.
Cluster amplification and sequencing
Cluster amplification of the templates was performed according to the manufacturer’s protocol (Illumina) using the Illumina cBot. Flow cells were sequenced on HiSeq 4000 Sequencing-by-Synthesis Kits, then analysed using RTA2.7.3
Host genetic data processing
Host genetic exome sequence data were processed using the Broad Institute sequencing pipeline by the Data Sciences Platform (Broad Institute). This was done in three steps: pre-processing (including reads mapping, alignment to a reference genome and data cleanup), variant discovery (including per-sample variant calling and joint genotyping), and variant filtering to produce callset ready for downstream genetic analysis, using Genome Analysis Toolkit (GATK) (detailed documentation at ).
Reduced representation bisulfite sequencing
Reduced representation bisulfite sequencing (RRBS) libraries were prepared for 221 biopsies and 228 blood samples as described previously70 with modifications detailed below. In brief, genomic DNA samples were quantified using a Quant-It dsDNA high sensitivity kit (ThermoFisher, Q33120) and normalized to a concentration of 10 ng/μl. A total of 100 ng of normalized genomic DNA was digested with MspI in a 20-μl reaction containing 1 μl MspI (20 U/μl) (NEB, R0106L) and 2 μl of 10× CutSmart Buffer (NEB, B7204S). MspI digestion reactions were then incubated at 37 °C for 2 h followed by a 15 min incubation at 65 °C.
Next, A-tailing reactions were performed by adding 1 μl dNTP mix (containing 10 mM dATP, 1 mM dCTP and 1 mM dGTP) (NEB, N0446S), 1 μl Klenow 3′-5′ exo– (NEB, M0212L) and 1 μl 10× CutSmart Buffer in a total reaction volume of 30 μl. A-tailing reactions were then incubated at 30 °C for 20 min, followed by 37 °C for 20 min, followed by 65 °C for 15 min.
Methylated Illumina sequencing adapters70 were then ligated to the A-tailed material (30 μl) by adding 1 μl 10× CutSmart Buffer, 5 μl 10 mM ATP (NEB, P0756S), 1 μl T4 DNA Ligase (2,000,000 U/ml) (NEB, M0202M) and 2 μl methylated adapters in a total reaction volume of 40 μl. Adaptor ligation reactions were then incubated at 16 °C overnight (16–20 h) followed by incubation at 65 °C for 15 min. Adaptor ligated material was purified using 1.2× volumes of Ampure XP according to the manufacturer’s recommended protocol (Beckman Coulter, A63881).
Following adaptor ligation, bisulfite conversion and subsequent sample purification were performed using the QIAGEN EpiTect kit according to the manufacturer’s recommended protocol designated for DNA extracted from FFPE tissues (QIAGEN, 59104). Two rounds of bisulfite conversion were performed yielding a total of 40 μl bisulfite-converted DNA.
In order to determine the minimum number of PCR cycles required for final library amplification, 50 μl PCR reactions containing 3 μl bisulfite-converted DNA, 5 μl 10× PfuTurbo Cx hotstart DNA polymerase buffer, 0.5 μl 100 mM dNTP (25 mM each dNTP) (Agilent, 200415), 0.5 μl Illumina TruSeq PCR primers (25 μM each primer)70 and 1 μl PfuTurbo Cx hotstart DNA polymerase (Agilent, 600412) were prepared. Reactions were then split equally into four separate tubes and thermocycled using the following conditions: denature at 95 °C for 2 min followed by X cycles of 95 °C for 30 s, 65 °C for 30 s, 72 °C for 45 s (where X number of cycles = 11, 13, 15 and 17), followed by a final extension at 72 °C for 7 min. PCR products were purified using 1.2× volumes of Ampure XP and analysed on an Agilent Bioanalyzer using a High Sensitivity DNA kit (Agilent, 5067-4626). Once the optimal number of PCR cycles was determined, 200-μl PCR reactions were prepared using 24 μl bisulfite-converted DNA, 20 μl 10× PfuTurbo Cx hotstart DNA polymerase buffer, 2 μl 100 mM dNTPs (25 mM each), 2 μl Illumina TruSeq PCR primers (25 μM each) and 4 μl PfuTurbo Cx hotstart DNA polymerase with the thermal cycling conditions listed above. PCR reactions were purified using 1.2× volumes of Ampure XP according to the manufacturer’s recommended protocol and analysed on an Agilent Bioanalyzer using a High Sensitivity DNA kit.
RRBS sequencing produced an average of 15.0M reads (s.d. 4.0M reads) over all 504 samples, with 448 (88.9%) samples exceeding 10M reads. Samples were analysed with Picard 2.9.4 using default parameters, resulting in a mean alignment rate to the human genome hg19 of 95.1%. Mean CpG coverage was 8.9× (s.d. 2.1%). As expected, 99.9% (s.d. 0.02%) of non-CpG bases and 49.8% (s.d. 2.8%) of CpG bases were converted.
Data handling
Informatics for microbial community sequencing data
For metagenomes and metatranscriptomes, sequencing reads from each sample in a pool were demultiplexed based on their associated barcode sequence using custom scripts. Up to one mismatch in the barcode was allowed provided it did not make assignment of the read to a different barcode possible. Barcode sequences were removed from the first read as were terminal Gs from the second read that may have been added by SMARTScribe during template switching.
Taxonomic and functional profiles were generated with the bioBakery meta’omics workflow71 v0.9.0 (). In brief, reads mapping to the human genome were first filtered out using KneadData 0.7.0. Taxonomic profiles of shotgun metagenomes were generated using MetaPhlAn272 v2.6.0, which uses a library of clade-specific markers to provide pan-microbial (bacterial, archaeal, viral, and eukaryotic) profiling (). Functional profiling was performed by HUMAnN273 v0.11.0 (). HUMAnN2 constructs a sample-specific reference database from the pangenomes of the subset of species detected in the sample by MetaPhlAn2 (pangenomes are precomputed representations of the open reading frames of a given species74). Sample reads are mapped against this database to quantify gene presence and abundance on a per-species basis. A translated search is then performed against a UniRef-based protein sequence catalogue75 (UniRef release 2014_07) for all reads that fail to map at the nucleotide level. The result are abundance profiles of gene families (UniRef90s), for both metagenomics and metatranscriptomics, stratified by each species contributing those genes, and which can then be summarized to higher-level gene groupings such as ECs or KOs.
Sample counts in Fig. 1b represent the numbers of raw products available. To ensure a reasonable read depth in each sample, only samples (metagenomes and metatranscriptomes) with at least 1 million reads (after human filtering) and at least one non-zero microbial abundance detected by MetaPhlAn2 were used in downstream analyses (Fig. 1c and later). In total, this was 1,595 metagenomic and 818 metatranscriptomic samples. Principal coordinates plots were generated with the cmdscale function in the R package stats. Visualizations were principally generated using ggplot276.
Species-level meta’omic functional profile summaries
Functional profiles per clade (typically species) were further quantified by summing the total sum-normalized stratified abundance attributed to each organism in the HUMAnN2 functional profiles from both metatranscriptomics and metagenomics. For metatranscriptomic analyses, the expression ratio for the species was then also defined as the ratio between these sums.
Metaproteomic gene family functional profiles
Gene family profiles were generated from metaproteomic peptides using UniRef90 identifiers by mapping peptide sequences to the Diamond-annotated reference in HUMAnN2 v0.11.0 with v0.8.22.8477. Each peptide was mapped to the UniRef90 with the highest per cent identity (minimum 90% match).
Statistical methods and association testing
PERMANOVA and Mantel tests
Omnibus testing was performed on Bray–Curtis dissimilarity matrices from MGX, MTX, MPX, and biopsy measurements. Functional profiles (MGX, MTX, and MPX) were first summarized to the KO level using HUMAnN2. Profiles were first normalized before calculation of dissimilarities. Dietary distance matrices were calculated by ordering the dietary intake frequencies from less to more frequent, assigning integers to these levels, and calculating the Manhattan distance.
Quantifications of covariation between measurement types in Fig. 1e were done using Mantel tests. To quantify cross-sectional (‘inter-individual’) covariation, we first produced an average profile for each subject by taking the feature-wise mean over all samples from the subject. Subject-subject dissimilarity matrices were then generated and compared using the mantel.rtest function in the R package ape4. To quantify longitudinal covariation, we first generated the complete sample–sample dissimilarity matrix, but only calculate the Mantel test statistic (the Pearson correlation between distances) from distances between samples from the same subject. Significance in this case was assessed using a permutation test with permutations limited within-subject.
Quantifications of variance explained in Fig. 1f were calculated using PERMANOVA with the adonis function in the R package Vegan78. Apart from the All row in Fig. 1f, the total variance explained by each variable was calculated independently of other variables (that is, as the sole variable in the model) to avoid issues related to variable ordering, and should thus be considered the total variance explainable by that variable. To account for the repeated measures present for all measurement types tested, relevant permutations were performed blocked within subject for variables that change over time (medication, biopsy location, inflammation status, and dysbiosis). Meanwhile, variables that were constant (or change slowly enough to be considered constant) across samples from the same subject (age, sex, body mass index (BMI), race, recruitment site, diagnosis, and disease location) were first permuted across subjects and samples were relabelled with the variable from their permuted subject. To determine the significance of models including inter-individual variance (the Subject and All rows), permutations were performed freely. For subjects with incomplete records for BMI, we imputed the mean BMI of the remaining population. The All row is the total variance explained when including all other variables in the model.
Differential microbiome feature abundance
Differential abundance (DA) analysis of all microbial measurement types (except for viruses, which were modelled as presence/absence binary features) were tested as follows. First, an appropriate transformation/normalization method was applied: arcsine square-root transformation for microbial taxonomic and functional relative abundances, log transformation (with pseudo count 1 for zero values) for metabolite profiles and protein abundances, and log transform with no pseudocount for expression ratios (non-finite values removed). Transformed abundances were then fit with the following per-feature linear mixed-effects model:
$$begin{array}{l},{rm{feature}} sim left({rm{intercept}}right)+{rm{diagnosis}}+{rm{diagnosis}}/{rm{dysbiosis}}+{rm{antibiotic}};{rm{use}}\ ,+{rm{consent}};{rm{age}}+left(1| {rm{recruitment}};{rm{site}}right)+left(1| {rm{subject}}right)end{array}$$
That is, in each per-feature multivariable model, recruitment sites and subjects were included as random effects to account for the correlations in the repeated measures (denoted by (1 | recruitment site) and (1 | subject), respectively) and the transformed abundance of each feature was modelled as a function of diagnosis (a categorical variable with non-IBD as the reference group) and dysbiosis state as a nested binary variable (with non-dysbiotic as reference) within each IBD phenotype (UC, CD, and non-IBD), while adjusting for consent age as a continuous covariate, and antibiotics as as binary covariate. Pearson’s residual values from the above linear mixed effects models were retained for use in subsequent analyses (see ‘Cross-measurement type interaction testing’).
Fitting was performed with the nlme package in R79 (using the lme function), where significance of the association was assessed using Wald’s test (except for viruses, where a logistic random effects model was considered with the glmer function from the lmer R package). Nominal P values were adjusted for multiple hypothesis testing with a target FDR of 0.25. In order to reduce the effect of zero-inflation in microbiome data, features with no variance or with >90% zeros were removed before fitting linear models. In addition, a variance filtering step was applied to remove features with very low variance. To further remove the effect of redundancy in KO gene family abundances (explainable by at most a single taxon), only features (both DNA and RNA) with low correlation with individual microbial abundances (Spearman correlation coefficient <0.6) were retained.
Differential host gene expression
Differentially expressed human genes between disease groups were quantified using a quasi-likelihood negative binomial generalized log-linear model (glmQLFit), implemented in the edgeR package in R80,81. Analysis was performed separately for each section of the intestine on genes with at least 2 CPM (counts per million) in 10 or more samples, with significance threshold FDR P < 0.05 and >1.5 log-fold change. Gene enrichment analysis was performed on differentially expressed genes against the KEGG database82 using a one-sided hypergeometric test in the package limma83.
Associations with host gene expression
Associations between host gene expression and biopsy taxonomic profiles were assessed using partial Spearman correlation, accounting for BMI, age at consent, sex and diagnosis. Association testing was performed for each biopsy location independently, as biopsy location was shown to heavily influence expression profiles (Figs. 1f, 4a, b). This simpler method was used rather than the more complex procedure outlined above for microbial measurement types since host gene expression, once filtered by biopsy location, do not have the same repeat measures problem as the microbial measurement types, allowing a simpler test.
Genetic associations
Genetic principal components for IBDMDB subjects as well as 1,000 Genomes subjects84 were calculated for a set of independent SNPs overlapping between the two data sets and pruned on the basis of linkage disequilibrium (LD). Pruning was first performed in HMP2 using the –indep-pairwise 1500 150 0.1 command in PLINK85 by calculating LD (r2) for each pair of SNPs within a window of 1,500 SNPs, removing one of a pair of SNPs if r2 > 0.1 and repeating this procedure by shifting the window 150 SNPs forward. We then used the 1,000 Genomes reference phase 3 version 5a data for 2,504 participants () to merge with the HMP2 pruned data, resulting in 7,227 overlapping independent SNPs. Using these, we performed genome-wide estimation of identity-by-descent allele sharing on the combined data set using the –genome function in PLINK, followed by calculation of genetic principal components using the –cluster–mds-plot function for the first two principal components (Fig. 1a).
For association analyses, we used first 20 genetic principal components as covariates, obtained from the same identity-by-descent sharing matrix using the –cluster–pca 20 function. We targeted associations in five loci that had strong previously reported associations with IBD and/or have been implicated in microbial interactions86,87,88. To avoid confounding by ancestry, we restricted the analysis to subjects of European ancestry, excluding eight subjects with exomes available from other ancestral backgrounds. When available, we used reported SNPs that had minor allele frequency of at least 5% and Hardy–Weinberg equilibrium P < 5 × 10−5. If not, we used close proxies (LD r2 < 0.8 in CEU population using 1,000 genomes phase 3 version 5 reference via (MST1, FUT2, IRGM, NKX2-3) or SNPs from the gnomAD browser at (PTGER4)).
We used the following linear mixed effect model with the SNP as a predictor variable, coded with an additive genetic model with the outcome as the arcsine-square root transformed microbial relative abundance measured from stool metagenomes. Age, sex, antibiotic and immunosuppressant use, and the first 20 genetic principal components (PCs) were fitted as covariates with subjects as the random effect:
$$begin{array}{l}{rm{taxon}} sim {rm{intercept}}+{rm{SNP}}+{rm{antibiotic}};{rm{use}}+{rm{sex}}+{rm{age}}\ ,+{rm{recruitment}};{rm{site}}+{rm{PC}}1-{rm{PC}}20+left(1| {rm{subject}}right)end{array}$$
Optimization was performed using the lme function (from the nlme R package), with P values calculated using the Wald test.
Associations between the rs1042712 SNP of the LCT lieu89 and self-reported milk intake from dietary recall forms were tested using the same mixed effect model. Reported dairy intake options were assigned the following numeric values for regression: 1 (‘yesterday, 3 or more times’), 2 (‘yesterday, 1 to 2 times’); 3 (‘within the past 2 to 3 days’), 4 (‘within the past 4 to 7 days’), and 5 (‘did not consume in last 7 days’).
Density ridgeline plots of differentially abundant features
To visualize the abundances of features that showed significantly different abundances by one of the tests above (Fig. 2, Extended Data Fig. 4), the bandwidth for kernel density estimation was selected independently for the portion of each feature above the detection limit (non-‘zero’) using the Sheather and Jones method90. Density estimates were scaled such that the maximum density for the plot spanned the distance between baselines for a given disease group. Samples below the detection limit are represented as barplots on the left, where a bar that spans the distance between baselines for a disease group represents 100% zeros. Density estimates were then additionally scaled by the fraction of non-zero samples such that relative differences in densities between groups with differing fractions of zeros are accurately represented. For both density estimates and fraction of zeros, samples were weighted by the inverse of the number of samples obtained from that subject, to avoid biasing estimates towards subjects with more densely sampled time series.
Dysbiosis analyses
Dysbiosis score
To identify samples with highly divergent (dysbiotic) metagenomic microbial compositions, as a complement to baseline disease diagnosis, we defined a dysbiosis score based on Bray–Curtis dissimilarities to non-IBD metagenomes. First, a ‘reference set’ of samples was constructed from non-IBD subjects by taking all samples after the 20th week after the subject’s first stool sample. This was chosen because a subset of the non-IBD subjects at the start of their respective time series may not yet have overcome any gastrointestinal symptoms that triggered the initial visit to a doctor, though these were ultimately not caused by IBD. The dysbiosis score of a given sample was then defined as the median Bray–Curtis dissimilarity to this reference sample set, excluding samples that came from the same subject (Fig. 2c).
To identify samples that were highly divergent from the reference set, we thresholded the dysbiosis score at the 90th percentile of this score for non-IBD samples. This therefore identifies samples with a feature configuration that has a less than 10% probability of occurring in a participant without IBD. By this measure, 272 metagenomes were classified as dysbiotic. Samples from participants with CD or UC were overrepresented in the dysbiotic set, with 24.3% and 11.6% of their samples classified as dysbiotic, respectively. As expected, these samples also tended to locate in the extremes of the taxonomic ordination based on metagenomes (Extended Data Fig. 3b, c). Dysbiosis was unevenly distributed among subjects (Extended Data Fig. 3d), with some subjects remaining dysbiotic for all or most of their time series, while others remained non-dysbiotic for their entire time series.
To lend additional support to the definition of dysbiosis (that is, as outliers by one type of microbiome profile), we tested the concordance between dysbiosis classifications made using the same statistical definition, but applied to metabolomic rather than taxonomic profiles. That is, we defined a metabolomic dysbiosis score as the median Bray–Curtis dissimilarity of one metabolomic profile to the non-IBD metabolomic profiles (after the 20th week), and defined the dysbiosis threshold as the 90th percentile of this distribution among non-IBD metabolomic profiles. We then compared these dysbiosis classifications with those from the nearest metagenomic sample (up to two weeks, see ‘Cross-measurement type temporal matching’) and found that dysbiotic samples identified by metagenomics were 4.6 times more likely to be dysbiotic by metabolomics (Fisher’s exact P = 5.9 × 10−9), showing that dysbiosis measurements are highly consistent across measurement types.
To test the sensitivity of the dysbiosis classification to the choice of reference data set, we also performed the dysbiosis classification using the HMP1-II stool samplesdix as the reference sample set instead of the non-IBD samples. The resulting dysbiosis scores (Extended Data Fig. 3e) were highly concordant (Spearman ρ = 0.86; P < 2.2 × 10−16), as were the dysbiosis classifications (odds ratio of 56; Fisher’s exact P < 2.2 × 10−16). This shows that, despite the inclusion of subjects with other conditions in the non-IBD group here, as well as large differences in measurement technologies between the data sets, the dysbiosis classification is highly robust. Furthermore, 43 out of 426 (10.1%) of non-IBD samples were classified as dysbiotic using the HMP1-II samples as reference, falling remarkably close to the 10% expected by the definition and showing that the enrichment of IBD samples in the dysbiotic set is not simply a consequence of the definition.
Dysbiosis durations and intervals
Samples of the dysbiosis durations and intervals were obtained by taking the difference in time between stool metagenomes in which the dysbiosis state changes, that is, the time from the first dysbiotic sample in an excursion into dysbiosis until the next non-dysbiotic sample was taken as one sample of the dysbiosis duration distribution. If the subject’s time series ended before this transition occurred, this resulted in a ‘censored’ duration or interval (Fig. 2e). Estimates of the durations of and time between dysbioses were then obtained from a censored maximum likelihood estimator for the mean of an exponential distribution. This incorporates the censored durations and intervals into the estimate to avoid underestimating these durations owing to limited observation times.
Association of dysbiosis with disease location
We tested for a relationship between the Montreal disease location classification in CD and periods of dysbiosis to ensure that dysbiosis was not simply detecting different disease locations. For this, an F-test of no significance was used with a Kenward–Roger approximation of degrees of freedom91 in a logistic random effects regression that models dysbiosis as the binary outcome with subjects as a random effect and disease location as covariate, as implemented in the function glmer in the R package lme4. Only individuals with CD were considered.
Temporal analyses
Power-law fits to Bray–Curtis dissimilarities
Power-law fits to species-level metagenomic, metatranscriptomic, and metabolite Bray–Curtis dissimilarities (Fig. 3a, Extended Data Fig. 5a) were performed by fitting a power-law curve with free intercept by least-squares using the neldermead function from the R package nloptr. Significance was assessed using an F-test to compare the fit model with a flat line. Significance of the difference of the fit between disease groups was also assessed using an F-test, comparing a model jointly fit to both disease groups with separate fits to each group.
Microbiome shifts
A microbiome ‘shift’ was defined as having occurred between two consecutive time points from the same person if the Bray–Curtis dissimilarity between their profiles was more likely to have come from a comparison between samples from different people rather than from the same person (Extended Data Fig. 5b). As an individual’s microbial profile naturally changes over time10,92, the Bray–Curtis threshold at which this occurs will increase with the time difference between samples. To determine these thresholds, kernel density estimates were generated for the distribution of Bray–Curtis dissimilarities between profiles from different individuals without IBD and between samples from the individuals without IBD at a range of time differences, using the density function in R. The point at which the inter-individual density estimate exceeded the intra-individual density estimate was then taken as the threshold to define a ‘shift’, with the additional constraint that this must be a monotonically increasing function of the time difference between samples (Fig. 3a). Metabolomic shifts were defined similarly, although owing to the more sparse temporal sampling of the metabolomics data (Extended Data Fig. 1) and lack of a strong upward trend in Bray–Curtis dissimilarities with time difference (Fig. 3a), only a single threshold was used based on the distribution of Bray–Curtis dissimilarities from comparisons within-subject over time in participants without IBD. Heatmaps of shift differences were generated using the R package pheatmap 1.0.1093.
Longitudinal multi-omic study design
Owing to the large variation in microbial profiles between people (Supplementary Fig. 2), with relatively smaller variation within subjects over time (Fig. 3a), longitudinal study designs have the potential to be higher-powered than purely cross-sectional studies, particularly in their ability to self-control individuals and to capture transitions between phenotypes (or after interventions) of interest94. Here, although some subjects remained in a dysbiotic state far longer than others (Extended Data Fig. 3d), the heterogeneity observed was enough to discover differences in measurement types other than where dysbiosis was defined. For example, subjects who had unusual, disease-associated microbiome taxonomic profiles also proved to have generally shifted serological and/or metabolomic profiles at corresponding time points. Noting these dysbiotic time points offered a complementary set of differences to what was visible cross-sectionally (Fig. 2).
Among microbially related measurements, metabolomics provided the most robust separation between disease and dysbiosis groups, possibly because it integrated a combination of host, microbial, and dietary differences (Fig. 1e, f). Thus, despite the challenges presented by untargeted metabolomics, such as unknown compound identification, the presence of redundant and background signals, and the complexity of the stool matrix, this measurement type often provides a robust characterization of subjects, their disease state, and individual small molecules that interface between host and microbiome. Conversely, the current state of viromics assays and reference databases makes this more challenging to work with, although the importance of the virome in microbial community dynamics95 will make this an extremely interesting feature space going forward.
All longitudinal microbial measurement types showed significant variation within two weeks (Fig. 3a, Extended Data Fig. 5a). This suggests that even higher sampling rates may be needed to catch relevant microbial variation, particularly before the onset of more severe clinical symptoms. Our sampling protocol also did not account for other potential sources of within-subject variation, such as transit time or the precise portion of each whole stool that was sampled. In this data set, we thus cannot distinguish between temporal and technical variation within subjects. To mitigate the higher costs associated with processing additional samples, a future study aiming to achieve higher temporal resolution might proceed in two phases, thanks to the coupling between data types (Fig. 1e): first, collect samples at a higher frequency, and process these with metagenomic or 16S sequencing. Then proceed with more expensive and detailed data generation only for samples taken specifically around periods of interest, such as periods of dysbiosis identified during the first stage. To this end, the sampling rate can also be tuned to target a particular probability of missing a dysbiosis period.
Integrative analyses
Lenient cross-measurement type temporal matching
For comparison between multiple measurement types, we first constructed sets of samples corresponding to the same biosample across data sets. However, exact matches were not always possible, for example, owing to specimen limitations during sample selection (see ‘Sample selection’) or to samples that failed quality control. In these cases, matching sample sets across data sets were created using nearby samples. During this process, a degree of leniency was allowed in the matching, allowing samples up to a given time difference (two or four weeks) apart to be ‘matched’. To perform this matching (sample numbers in Fig. 1c and Extended Data Fig. 1b), we used the following algorithm.
For a given set of measurement types to be matched for a particular subject, find the first time window in which all measurement types have at least one produced sample. Next, within this window, find the time point with the most measurement types produced; in the event of a tie, select earlier time points. Finally, for each data set, select the nearest sample to this target time point that is within the time window, breaking ties towards earlier time points. This set of selected samples comprises one ‘matched’ sample. For each data set, all samples up to and including the later of the selected sample or the target time point are then disregarded from future consideration (and thus any sample will be included in at most only one matched time point). This process is repeated for each subject until no such window exists.
Cross-measurement type interaction testing
Significant associations between features from multiple measurement types were identified using two different models: an ‘unadjusted’ model of associations that are mainly due to dysbiosis, and an ‘adjusted’ model that emphasizes associations in addition to those that are dysbiosis-linked. Associations in both cases incorporated features from ten data sets: metagenomic species, species-level transcription ratios, functional profiles at the EC level (MGX, MTX and MPX), metabolites, host transcription (rectal and ileal separately), serology and faecal calprotectin.
To detect adjusted associations, we first obtained residuals of features from the above data types fit to a mixed-effects model including subjects as random effects as above for the differential abundance testing (or a simple linear model without the random effects when only baseline samples were used) and adjusting for age, sex, diagnosis, dysbiosis status, antibiotic and immunosuppressant use, and bowel surgery status. Residuals from subjects with fewer than four samples in their time series for the measurement type were ignored, and for measurements with no longitudinal samples (for example, serology and host transcriptomics measurements), the residualization was repeated with the first available samples using a simple linear model without random effects. This allowed the identification of significant (FDR P < 0.05 for most measurement types, P < 0.25 for serology) Spearman associations using HAllA 0.8.17 (hierarchical all-against-all association testing, , Supplementary Table 35). As subject-specific random effects and covariate effects were removed from these residuals, the resulting correlations are likely to be independent of all sources of inter-individual variation as well as confounding effects due to the covariates. See Fig. 4c and Extended Data Figs. 7–9 for summary visualizations of these results. Similarly, unadjusted associations were identified using the same procedure, but without including dysbiosis as a covariate (Supplementary Table 36). Network visualization was done using Cytoscape96 3.6.0.
Reporting summary
Further information on research design is available in the Nature Research Reporting Summary linked to this paper.
[ad_2]