[ad_1]
Pour les cinéphiles, Space Jam était un film de comédie de 1996 opposant le personnage de dessin animé Bugs Bunny et le basketteur Michael Jordan aux extraterrestres animés. Pour le neuroscientifique Ed Lein, c’était le nom d’une réunion sur le thème de la bioinformatique – une sorte de «hackathon».
En avril, environ 40 biologistes de l'informatique et de la transcription se sont rendus à l'Institut Allen pour la science du cerveau à Seattle, Washington, où travaille Lein. Ils sont venus pour le café, la codification et un objectif commun: déterminer les forces, les faiblesses et les défis analytiques de l’ensemble d’outils méthodologiques croissant appelé in situ transcriptomique (ou spatiale).
In situ La transcriptomique est un concentré de technologies – les méthodes incluent MERFISH, seqFISH +, STARmap et FISSEQ – pour cartographier les modèles d'expression génique de cellules dans leur contexte tissulaire. Certains reposent sur l'hybridation – la capacité des sondes d'acide nucléique courtes à trouver leur complément dans l'environnement cellulaire surpeuplé – tandis que d'autres sont basés sur le séquençage de l'ADN. Mais tous produisent des données conceptuellement similaires – des valeurs d’expression de gènes correspondant à la X et y les coordonnées d'une cellule.
De telles données peuvent révéler des relations intercellulaires qui pourraient autrement être négligées, telles que les cellules avec lesquelles elles parlent, et leur position par rapport aux caractéristiques structurelles et aux cellules d'intérêt. Comme le dit Aviv Regev, biologiste des systèmes et des systèmes, et co-président fondateur du projet Human Cell Atlas (HCA) du Broad Institute of MIT et de Harvard à Cambridge, dans le Massachusetts, dites-moi qui est votre voisin et moi. Je vais vous dire qui vous êtes. "
Mais la croissance de ce domaine est si rapide que les chercheurs pourraient avoir du mal à décider des méthodes à utiliser. Et la pléthore d'algorithmes d'analyse de données, de pipelines et de formats de fichiers peut rendre difficile l'analyse et la comparaison de données. «L’état du terrain a été caractérisé par un développement technologique effréné», déclare Lein.
Avec le financement de l'organisation philanthropique Chan Zuckerberg Initiative (CZI) et sous les auspices de la HCA, Lein et d'autres ont formé un consortium de recherche en 2017 pour comparer les différentes méthodes, SpaceTx – abréviation de transcriptomique spatiale. Parallèlement, les programmeurs de la CZI ont commencé à créer un outil unifié d’analyse des données et un format de fichier, appelé Starfish, afin de faire progresser les efforts de la CHA et d’aider la communauté plus large de la biologie transcriptionnelle. (Le nom "est un peu une blague", explique Jeremy Freeman, qui dirige les efforts de biologie computationnelle au CZI de Redwood City, en Californie. De nombreuses méthodes spatiales reposent sur FISH, ou la fluorescence in situ hybridation. En programmation, un astérisque ou une étoile indique un caractère générique. "La blague est qu’ils sont tous" quelque chose-poisson ".")
Starfish est une suite logicielle open-source capable de lire, d’enregistrer et de supprimer le bruit des images, de localiser les taches et d’identifier les molécules d’ARN qu’elles représentent dans neuf stratégies expérimentales différentes, deux autres étant en cours de développement. Selon Lein, le Space Jam a été conçu pour réunir les développeurs et les utilisateurs, les spécialistes de la transcriptomique spatiale eux-mêmes, dans le but de discuter, de dépanner et de faire progresser leurs méthodes. Ce faisant, l'équipe a exposé les différences subtiles qui peuvent dérouter ceux qui veulent, par exemple, comparer des données entre expériences. Mais cela a également fourni un modèle sur la manière de naviguer dans une technologie en pleine croissance.
In situ transcriptomique
Les chercheurs qui étudient l'expression des gènes l'ont généralement fait en extrayant l'ARN d'un morceau de tissu, puis en l'analysant dans son intégralité. Au cours des dix dernières années, les méthodes monocellulaires telles que Drop-seq ont permis aux chercheurs de sonder les différences entre les cellules au détriment des détails spatiaux.
C'est là que in situ la transcriptomique entre en jeu. Ces techniques utilisent principalement la microscopie à fluorescence et le séquençage de l'ADN pour révéler la présence et l'abondance de molécules d'ARN dans les cellules des tissus eux-mêmes. À partir de là, les chercheurs peuvent déterminer les types de cellules présentes, leur organisation spatiale et leurs relations les unes avec les autres.
C’est comme une sélection de desserts fruités, dit Regev. «Si toute la génomique en vrac est le smoothie aux fruits, la génomique unicellulaire est la salade de fruits, et la génomique spatiale est la tarte aux fruits», explique-t-elle. "Si vous regardez une tarte aux fruits du haut, tous les fruits sont organisés dans ces très beaux modèles."
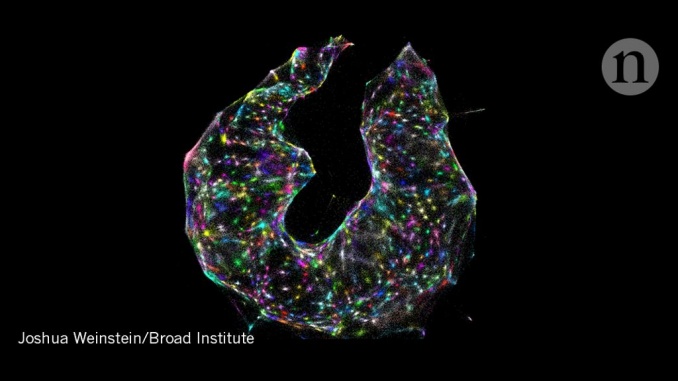
Selon la méthode utilisée, ces données peuvent ressembler à des étoiles dans un ciel noir, ou à des œuvres d'art colorées. Une étude menée par Simone Codeluppi, informaticienne en bioimage dans le laboratoire de Sten Linnarsson à l'Institut Karolinska de Stockholm, a par exemple utilisé une variante cyclique de FISH à une seule molécule, appelée osmFISH (prononcé «poisson impressionnant»), du cortex somatosensoriel de souris. Le résultat était une image des cellules colorées sur la base de leurs modèles d'expression génique, une image qui rappelle un vitrail..
Mais de telles données peuvent aussi révéler des idées. David Rowitch, neurobiologiste et médecin à l'Université de Cambridge, a utilisé une méthode appelée RNAscope pour étudier la diversité spatiale et l'organisation des astrocytes dans le cerveau de souris.. Rowitch a constaté que les astrocytes «adoptent des schémas de couche dans le cortex similaires aux neurones, mais hors de registre». Long Cai, qui étudie la biologie unicellulaire à la California Institute of Technology de Pasadena, et son équipe ont utilisé une stratégie appelée seqFISH + pour identifier les transcrits codant pour les protéines en interaction à la surface des cellules adjacentes..
Fournir de la clarté
SeqFISH + et RNAscope reposent tous deux sur l'hybridation acide nucléique; ils exploitent de courtes molécules marquées par fluorescence pour éclairer leurs séquences cibles dans la cellule. D’autres méthodes utilisent le séquençage de l’ADN ou même la spectrométrie de masse (voir «Soupe à l’alphabet»).
Plus d'une douzaine de méthodes de transcriptomique spatiale ont été décrites, dont six en 2019–. Ils diffèrent par le nombre d'ARN qu'ils peuvent détecter, leur résolution spatiale et le nombre de cellules qu'ils peuvent détecter, mais ils fournissent tous les détails de localisation spatiale que la transcriptomique unicellulaire ne peut pas. Mais les méthodes spatiales ont aussi des inconvénients, dit Regev. La microscopie, par exemple, est lente (impliquant parfois plusieurs semaines d’imagerie en continu), coûteuse et techniquement exigeante. De nombreuses méthodes ne peuvent accéder qu’à une fraction prédéfinie du transcriptome cellulaire et des considérations pratiques peuvent limiter le nombre de cellules pouvant être sondées.
En essayant de choisir la bonne méthode pour leur travail, les chercheurs pourraient être submergés. SpaceTx vise à fournir une certaine clarté.
Le projet a été financé dans le cadre des quelque 100 millions de dollars US dépensés par la CZI au cours des deux dernières années et demie pour le HCA et les projets auxiliaires, a déclaré un porte-parole de la CZI. Chaque équipe – il y avait au total 19 membres – a appliqué sa propre méthode à des échantillons identiques de cerveau humain et de cerveau de souris, qui ont été préparés à l'Institut Allen. Maintenant, Lein et ses collègues, ainsi que la communauté plus large de la biologie computationnelle, analysent les chiffres pour voir comment les méthodes se comparent et quelle est la meilleure solution pour un ensemble de circonstances donné.
"C'est en fait assez inhabituel", dit Lein. Normalement, les chercheurs travaillent à développer la meilleure méthode, à publier et à passer à autre chose. Mais avec SpaceTx, "nous essayons de rassembler tout le monde et de dire que ces méthodes sont toutes utiles, mais nous devons comprendre à quoi sert chaque méthode et comment elles se comparent quantitativement les unes aux autres."
Mais cela pose un problème de calcul, car différentes méthodes produisent différents types de données. Certaines méthodes basées sur l'hybridation, par exemple, attribuent à chaque transcript une couleur différente, tandis que d'autres utilisent plusieurs couleurs comme code à barres. Certains laboratoires identifient les ARN en recherchant les points fluorescents dans chaque image, puis en surveillant leur intensité entre les cycles d'imagerie, tandis que d'autres mesurent l'intensité à chaque pixel, en corrélant ces valeurs avec la liste des codes à barres possibles pour déterminer la présence ou non d'ARN. La manière dont ces images sont organisées sur le disque et les ‘métadonnées’ utilisées pour les annoter peuvent également varier.
Matthew Green, bioinformaticien à l'European Bioinformatics Institute de Hinxton, au Royaume-Uni, estime que de telles incompatibilités peuvent entraver la recherche. Même s’ils ne le font pas, les chercheurs ont souvent du mal à installer le logiciel d’analyse de leurs collègues (en raison des besoins informatiques complexes et des dépendances qu’un tel logiciel entraîne). Et le volume de données généré par les études spatiales peut être intimidant – la plate-forme osmFISH automatisée de Linnarsson génère 2 téraoctets d’images par jour, dit-il; pour SpaceTx, son équipe a produit environ 25 téraoctets.
Space Jam
Une équipe de biologistes informaticiens et d'ingénieurs en logiciel dirigée par Deep Ganguli et Ambrose Carr au CZI a pour objectif de créer un format de fichier et un pipeline standard pour in situ analyse du transcriptome – un moyen de combiner différentes méthodes de calcul et de laboratoire humide, que ce soit sur un ordinateur portable ou dans le cloud. L'équipe est même allée sur la route, visitant des laboratoires et parlant à des bioinformaticiens pour comprendre leurs flux de travail. «Au moins un des étudiants diplômés de l’un des laboratoires a déclaré à Deep:« C’est tellement merveilleux, car personne n’avait jamais regardé mon code », dit Freeman.
Le hackathon d’avril a donné l’occasion à l’équipe Starfish de laisser les biologistes tester le logiciel, offrant ainsi aux chercheurs et aux codeurs l’occasion de tirer des leçons mutuellement de leurs expériences, plutôt que par le biais de rapports de bogues sur la plateforme de partage de code GitHub.
«Nous avons pu aider tous ces gens à mettre en place leurs pipelines de traitement de données dans Starfish pour les aider dans leurs efforts scientifiques», a déclaré Justin Kiggins, responsable du développement de Starfish à la CZI. "Et cela a donné à notre équipe un aperçu critique des lacunes et des défis."
Matt Cai, étudiant diplômé en bio-ingénierie à l'Université de Californie à San Diego, qui a mis au point une méthode appelée DARTFISH, affirme qu'il avait deux objectifs lors du hackathon de Seattle: échanger des idées avec d'autres groupes de transcriptomique spatiale et se familiariser avec Étoile de mer. «Nous avons nos propres méthodes d’analyse internes, mais elles ne sont pas écrites de manière à ce qu’elles soient faciles à utiliser», dit-il. "Starfish est écrit pour la communauté scientifique."
Pour Green, la réunion ne ressemblait à aucune autre. Bien qu’il ait assisté à plusieurs conférences au fil des ans, «je n’ai jamais assisté à une première réunion», dit-il. «Chaque conversation était littéralement un échange d'informations massif. Et c'était assez excitant.
Chaque équipe a réussi à convertir un échantillon de données au format Starfish, explique Lein, et la génération de données est en cours. Mais le logiciel lui-même reste un travail en cours. Aleksandra Tarkowska, programmeuse au Wellcome Sanger Institute de Hinxton, au Royaume-Uni, a déclaré ne pas être en mesure de convertir ses ensembles de données au format Starfish et d'aligner différents champs de vision dans une image unifiée, en raison de «la complexité des données». Et Nico Pierson, un ingénieur en logiciel du laboratoire de Long Cai, a signalé des problèmes avec le «décodeur ponctuel» du logiciel, l’algorithme qui fait correspondre les modèles de fluorescence aux codes à barres, car il était incapable de gérer la densité des données seqFISH +. "Avec nos données, l'efficacité est très faible, elle ne représente probablement que 10%", dit-il.
Néanmoins, les participants ont félicité l’événement d’avoir fait parler les programmeurs et les biologistes. Les programmeurs sont repartis avec une pile de rapports de bugs et de demandes de fonctionnalités, dont certaines pourraient être résolues sur-le-champ. Et les chercheurs sont retournés dans leurs laboratoires armés de nouvelles et meilleures idées pour l'analyse de données. Codeluppi, par exemple, a découvert une stratégie de «segmentation» permettant d’identifier par calcul les limites des cellules dans ses données d’image, en particulier pour les cellules de petit volume.
Matt Cai explique que son laboratoire utilise désormais régulièrement Starfish avec son propre pipeline de calcul pour comparer les performances. Mais d'autres pourraient être réticents à abandonner les canalisations internes qu'ils ont si minutieusement conçues. Starfish pourrait donc trouver sa plus grande adoption parmi les laboratoires qui tentent de mettre en œuvre les méthodes développées par d’autres.
"Avec toutes les approches différentes, il est très utile d'avoir quelque chose qui puisse les lier ensemble dans un format informatique, et Starfish permettra, je pense, que cela se produise", déclare Abbas Rizvi, neuroscientifique moléculaire au Zuckerman Mind Brain Behavior Institute de Université Columbia à New York. Rizvi est un membre du projet HCA qui construit un atlas de la moelle épinière humaine en utilisant, en partie, des méthodes spatiales.
«Cela me rappelle les premiers stades de la transcriptomique unicellulaire», dit-il. «C'était assez difficile de faire en sorte que les expériences fonctionnent, mais il était également passionnant d'examiner les données et d'essayer de trouver des moyens d'en extraire un sens réel. Et c’est là que je vois le terrain en ce moment.
[ad_2]