[ad_1]
Chaque cellule humaine contient tellement d’ADN – environ 2 mètres si elle est étendue – qu’elle doit être étroitement entourée de protéines histones spécialisées pour former des structures en forme de spool appelées nucléosomes. Les nucléosomes peuvent ensuite être regroupés dans des brins denses appelés chromatine, dans lesquels l'ADN est inaccessible, et doivent être décompressés pour que l'ADN soit accessible à des fins de transcription ou de réplication. La conversion dynamique entre les états de la chromatine inaccessibles et accessibles est dirigée par des complexes protéiques qui écrivent et lisent des marques chimiques sur la chromatine appelées modifications épigénétiques. , Xue et al. décrivent la structure liée aux nucléosomes des membres de la famille des protéines MLL: complexes qui ajoutent des groupes méthyle aux protéines histones. Les nouvelles structures montrent comment ces complexes protéiques écrivent et lisent des modifications épigénétiques.
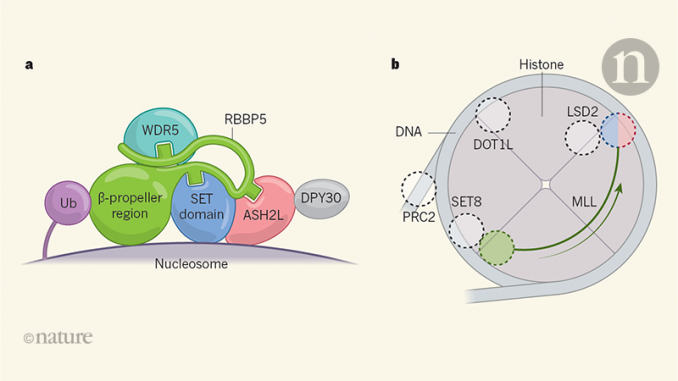
Les complexes MLL sont constitués de cinq protéines centrales, y compris une protéine MLL, qui possède un domaine SET qui contient le site catalytique du complexe. L’organisation du complexe MLL humain est conforme à celle observée dans les analyses de structures de l’équivalent levure, et méthyle le même résidu d'acide aminé de lysine (lysine 4, abrégé en K4) de l'histone H3 (H3K4). Cela implique qu'une version de ce complexe a joué ce rôle au cours d'une longue période d'évolution. Une autre partie du complexe MLL (la protéine RBBP5) lit un groupe d'ubiquitine sur le résidu de lysine 120 de l'histone H2B (H2BK120ub), qui favorise l'activité de méthylation de H3K4..
En utilisant une technique appelée microscopie cryo-électronique (cryo-EM), Xue et al. déterminé les structures de deux complexes MLL (l’un contenant MLL1 et l’autre contenant MLL3) liés à leur nucléosome cible. Les auteurs ont mené des analyses structurelles et biochimiques pour étayer leur modèle de fonctionnement du complexe MLL. Globalement, ils montrent que, à la manière d’un verrou et d’une clé, plusieurs parties distinctes du complexe doivent être reliées ensemble dans une configuration particulière pour «activer» l’activité de méthylation du complexe (Fig. 1a).
Dans le complexe MLL, la région autrement non structurée de RBBP5 appelée région post-β-hélice devient ordonnée et aligne et active la sous-unité de méthylation (figure 1a). En outre, les structures ont également révélé que le domaine β-propeller de la sous-unité RBBP5 établit un contact majeur avec le nucléosome et avec la marque H2BK120ub, qui agit également pour stabiliser et activer davantage le complexe. Ainsi, les sous-unités protéiques doivent être organisées exactement dans le complexe pour «déverrouiller» la chromatine.
La structure établie par Xue et al. révèle que le complexe MLL reconnaît à la surface du nucléosome des caractéristiques différentes de celles reconnues par d'autres complexes caractérisés de protéines modifiant la chromatine (Fig. 1b). Ces complexes incluent DOT1L,, SET8 et LSD2, qui lient chacun différents sites sur la face du nucléosome, ainsi que le complexe PRC2, qui se lie au bord de son substrat nucléosome. Les différences de sites de liaison entre ces complexes peuvent être attribuées au fait qu'ils doivent chacun accéder à différents résidus cibles tout en lisant simultanément d'autres marques épigénétiques particulières. Par exemple, alors que le complexe MLL se lie simultanément à H3K4 et à la marque H2BK120ub, PRC2 doit se lier à la lysine 27 dans une histone H3 (H3K27) avec son site actif tout en se liant à un résidu K27 dans une autre molécule H3 déjà modifiée par la triméthylation.
Les connaissances antérieures sur les bases structurelles et mécanistiques de la régulation de la chromatine ont nécessairement été limitées par les capacités des méthodologies existantes. La cristallographie aux rayons X a souvent été utilisée pour cibler des protéines uniques, atteignant généralement une résolution de 2,5 ångströms ou supérieure. Plus récemment, l’analyse cryo-EM a permis de visualiser des complexes beaucoup plus grands, mais à une résolution un peu inférieure (supérieure à 4 Å).
La cristallographie aux rayons X nécessite généralement que la structure de la molécule étudiée soit cohérente dans l’ensemble de l’échantillon. Les structures qui sont déterminées en utilisant EM peuvent souvent exister dans diverses conformations au sein du même échantillon. En cristallographie aux rayons X et en ME, un mythe courant est que les parties les plus importantes d’une structure examinée ont tendance à être les plus difficiles à définir, et que les deux techniques sont sensibles au fait que certaines parties de protéines sont intrinsèquement mieux ordonnées que les autres. autres. Cependant, dans la cryo-EM, les interactions moléculaires faibles qui existent à divers degrés dans un échantillon peuvent être stabilisées, par exemple en introduisant des réticulations covalentes, permettant la détection de multiples conformations possibles de protéines ou de complexes. Ainsi, l’analyse à une seule particule peut être utilisée pour exploiter les différences entre différentes conformations d’une protéine ou d’un complexe afin de mieux comprendre sa biologie.
Les structures de complexes multi-protéines MLL liées à des nucléosomes par Xue et ses collègues révèlent comment une série d’interactions faibles au sein du complexe et entre le complexe et le nucléosome agissent en synergie pour activer l’activité de la sous-unité enzymatique du complexe. Ainsi, bien que la présence de la marque H2BK120ub n’affecte pas sensiblement l’affinité du complexe MLL pour le résidu H3K4, elle double l’activité de méthylation du complexe.
L'existence de multiples conformations structurelles d'une protéine ou d'un complexe a été une complication déconcertante dans les études EM. Cependant, la mise au point d'outils d'analyse plus sophistiqués permettant d'interpréter les données sur la cryo-EM signifie que différentes conformations peuvent être décrites et examinées afin de révéler une richesse d'informations biologiques. Xue et al. décrivent plusieurs configurations du complexe MLL qui interagissent avec l’ubiquitine et le nucléosome de différentes manières. Les études futures devraient révéler la pertinence biologique du mode de liaison dynamique de ce complexe.
[ad_2]